Система учета научной деятельности (ASSA) |


|
Лаборатория компьютерной протеомики (т.12)Отдел системной биологии
Научные результаты Сотрудники О Подразделении 1. Основное направление исследований Изучение структурно-функциональной организации белков
2. Задачи, решаемые в рамках базового бюджетного проекта на данном этапе: В рамках проекта VI.61.1.2. «Компьютерно-экспериментальное исследование и моделирование структурно-функциональной организации и эволюции генных сетей многоклеточных и одноклеточных организмов» решаются задачи по направлению компьютерный анализ и моделирование структурно-функциональной организации белков, включая: (1) совершенствование существующих и разработка новых алгоритмов компьютерной структурно-функциональной аннотации белков (методы молекулярного моделирования, методы предсказания сайтов связывания малых химических соединений, сайтов белок-белок взаимодействий, активных центров и методы предсказания термодинамической стабильности белков); (2) исследование взаимосвязи между структурой белков и их функциональными, аллергенными и иммуногенными свойствами на основе анализа конформационных пептидов, моделирующих белковые молекулярные поверхности; (3) изучение закономерностей структурной организации функциональных сайтов белков; (4) анализ закономерностей структурной организации ассоциативных сетей белок-белок взаимодействий, связанных с заданной клеточной/системной функцией, биологическим процессом или заболеванием; (5) разработка новых подходов для поиска фармакологических мишеней и дизайна молекулярных соединений – кандидатов для лекарственных препаратов и проведение, на этой основе, компьютерного дизайна/скрининга ряда химических соединений, обладающих потенциальным противовирусным и антибактериальным действием; (6) изучение влияния мутаций на изменение термостабильности белков, функционирующих в составе изучаемых генных сетей, с использованием методов молекулярной динамики. (7) создание интегрированных информационных хранилищ данных по структуре, функции и эволюции белков. С помощью интегрированных подходов будет проводиться анализ метапротеомов бактериальных сообществ.
3. Прикладные разработки Разработана программно-информационная система Protein Structure Discovery (Иванисенко и др., 2011), предназначенная для решения широкого круга задач компьютерной протеомики, включая предсказание функции, структуры и иммунологических свойств белков. Специально созданный раздел системы позволяет оценивать количественные и качественные эффекты мутаций на структурные и функциональные свойства белков. Всего в систему интегрировано более 20 различных программ, в том числе база данных PDBSite (Ivanisenko et al, 2005), содержащая информацию о пространственных структурах более 100000 функциональных сайтов белков, программа распознования функциональных сайтов в пространственных структурах белков PDBSiteScan (Ivanisenko et al, 2004), программа количественного анализа взаимосвязи структура-активность белков WebProAnalyst (Ivanisenko et al, 2005), система SitEx (Medvedeva et, al, 2012), предназначенная для решения задач по анализу соотношения экзон-интронной структуры генов и структурно-функциональной организации, кодируемых ими белков; программа Allpred (Bragin et al, 2013), предназначенная для предсказания аллергенности белков с использованием конформационных пептидов и другие. Protein Structure Discovery имеет Вэб-интерфейс и доступна пользователям через Интернет (http://www-bionet.sscc.ru/psd).
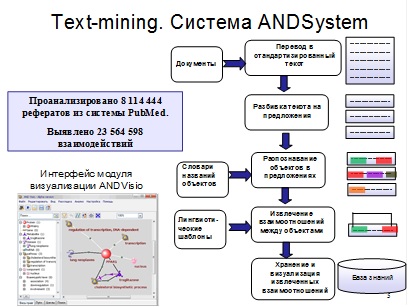
4. Иллюстрированное описание лучших результатов, полученных подразделением за последние 5 лет. Общий объем текста на одну лабораторию (сектор) – не более 4000 знаков с пробелами, количество иллюстраций с подписями: от одной до – пяти. Рекомендуется, чтобы общий объем текста с иллюстрациями не занимал более 5 страниц в формате Word (для текста шрифт Nimes New Roman, фонт № 12, через 1 интервал). 1) Разработана компьютерная система ANDSystem автоматического анализа текстов научных публикаций и баз данных (Demenkov et al, 2011). Система содержит модуль извлечения знаний из текстов и баз данных, базу знаний ANDCell и программу ANDVisio, обеспечивающую доступ к базе знаний и визуализацию ассоциативных генных сетей (рис. 1). Система позволяет автоматически извлекать из текстов информацию о широком круге взаимодействий и взаимоотношений между белками, генами, микроРНК, метаболитами, болезнями и биологическими процессами, включая физические взаимодействия, регуляторные отношения, транспорт и др.
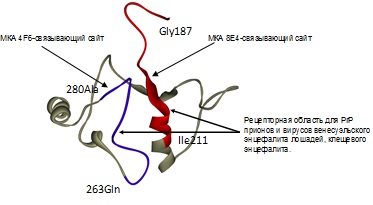
Рисунок 1. Схематичное представление системы ANDSystem автоматического извлечения знаний из текстов научных публикаций и биологических баз данных. 2) Найден гипотетический рецепторный домен служащий рецептором для проникновения прионов и вирусов в клетку (Малыгин и др. 2009). Проведено компьютерное моделирование пространственной структуры ламининсвязывающего белка человека (LBP). Показано, что разрывные участки последовательности C-концевого домена LBP, взаимодействующие с гликопротеином Е вируса венесуэльского энцефалита лошадей (VEE) и вируса клещевого энцефалита (TBE), оказываются сближенными в пространственной структуре и образуют рецепторный домен. Кроме того, анализ показал, что данный домен служит рецептором для проникновения прионов в клетку, таким образом, существует общий путь проникновения для вирусов и прионов (рис. 2).
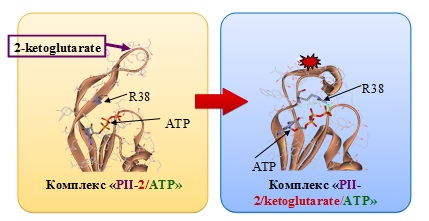
3) Компьютерный анализ белка PII Mycobacterium tuberculosis, являющегося важнейшим регулятором метаболизма азота, сигнальной трансдукции и мембранного транспорта в микобактерии, проведенный с использованием программы PDBSiteScan и молекулярной динамики, выявил в данном белке сайты связывания 2-кетоглютората (рис. 3). Молекулярное моделирование показало, что связывание 2-кетоглютората приводит к перестройке белка PII и повышению его сродства к АТФ. Экспериментальная проверка на приборе BIOCORE подтвердила результаты компьютерного анализа (Bandyopadhyay et al., The Journal of Biochemistry, 2010). Таким образом, показано, что 2-кетоглюторат действует как аллостерический регулятор связывания PII с ATФ.
Рисунок 3. Графическое изображение фрагмента модели пространственной структуры белка PII в комплексе с АТФ и 2-кетоглютарат.
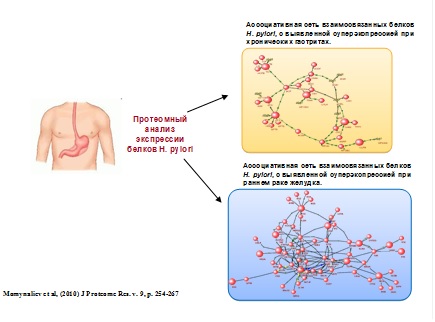
4) На основе анализа экспериментальных данных протеомных исследований, полученных в Институте физико-химической медицины (Москва) проведена реконструкция и анализ сетей молекулярно-генетических взаимодействий белков Helicobacter pylori, дифференциально экспрессирующихся в различных штаммах, выделенных у пациентов с хроническими гастритами и опухолями желудка (рис. 4). Показано, что различия в экспрессии белков H. pylori могут быть связаны с адаптацией этого микроорганизма к различным условиям внешней среды, обусловленными морфологическими отличиями различных участков желудка, подверженных гистологическим и морфологическим изменениям в ходе патологических процессов (Momynaliev et al, 2010). Для реконструкции ассоциативных сетей использовалась система автоматического анализа текстов научных публикаций и баз данных ANDSystem.
Рисунок 4. Ассоциативные сети взаимосвязанных белков H. pylori, идентифицированных в протеомных экспериментах, построенные с использованием системы автоматического анализа научных текстов ANDSystem.
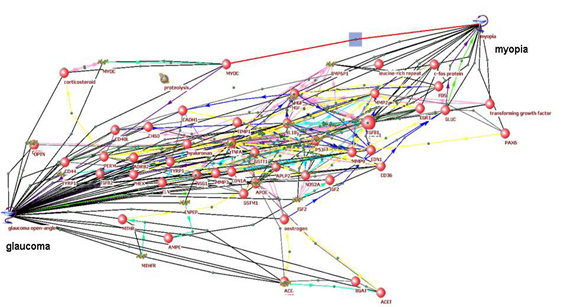
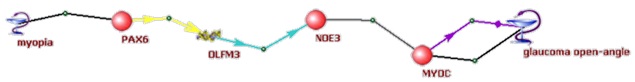
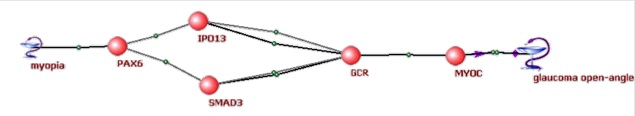
5) С использованием компьютерной системы ANDSystem проведена реконструкция и анализ ассоциативной сети, описывающей потенциальные молекулярные механизмы взаимосвязи миопии и открытоугольной глаукомы (Подколодная и др., 2010). Показано, что ассоциативная сеть содержит более 200 различных генов, ассоциированных с этими заболеваниями, и более чем 2000 молекулярно-генетических взаимодействий между данными генами. Редукция сети и дальнейший анализ представленных в ней молекулярно-генетических путей позволили выявить ряд потенциальных генов-кандидатов для генотипирования одновременно миопии и открытоугольной глаукомы (рис. 5).
А
Б
В Рисунок 5. Молекулярно-генетические пути, ассоциированные с открытоугольной глаукомой и миопией. (А) Наиболее значимые взаимодействия между генами и белками человека, ассоциированными с открытоугольной глаукомой и миопией; (Б) пути, направленные от миопии к глаукоме; (В) пути, с нечетко определенным типом и направлением взаимодействий.
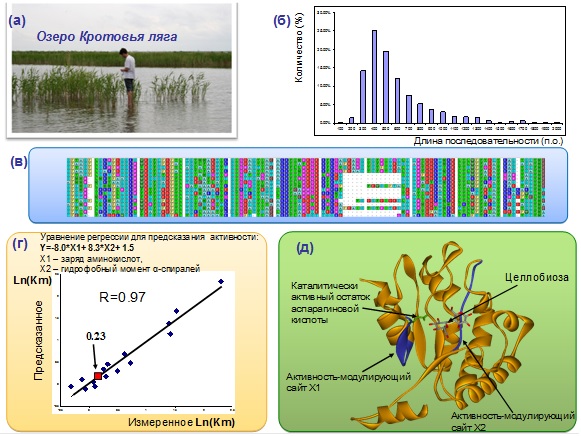
6) Разработан (совместно с Институтом физико-химической биологии ФМБА) новый подход к поиску белков, перспективных для биотехнологии (Иванисенко и др., 2012). Подход основан на параллельном высокопроизводительном секвенировании метагеномов и компьютерном анализе: сборке коротких секвенированных фрагментов в протяженные геномные последовательности (контиги); поиске в них открытых рамок считывания; функциональной классификации кодируемых ими ферментов и предсказании количественных величин их функциональной активности. На основе анализа результатов секвенирования метагенома микробных сообществ озера «Кротовья ляга», выполненного на платформе SOLiD, идентифицирован белок гликозидаза (фермент деградации целлюлозы) с высокой функциональной активностью (рис. 6).
Рисунок 6. Новый подход к поиску белков, перспективных для биотехнологии, основанный на параллельном высокопроизводительном секвенировании метагеномов и компьютерном анализе. (а) Озеро «Кротовья ляга» - источник генетического материала для метагеномного секвенирования микробных сообществ; (б) распределение длин нуклеотидных последовательностей, собранных в контиги; (в) фрагмент множественного выравнивания белковых последовательностей, потенциальных гликозидаз, кодируемых открытыми рамками считывания; (г) предсказание значения количественной величины константы Михаэлиса для потенциальной гликозидазы в реакции расщепления целлобиозы; (д) реконструированная модель пространственной структуры гликозидазы, обладающей повышенной активностью расщепления целлобиозы.
7) Предсказана мишень действия соединения NIH415032 (рис. 7), обладающего согласно экспериментальным данным противотуберкулезной активностью (Gahoi et al, 2013). С использованием программы PDBSiteScan и методов виртуального скрининга показано, что миколилтрансфераза (антиген 85) M. tuberculosis, один из ключевых ферментов, участвующих в биосинтезе микобатериальных клеточных стенок, является потенциальной мишенью для соединения NIH415032, для которого ранее было показана противотуберкулезная активность, но мишень его действия была не известна (Gahoi et al, 2013).
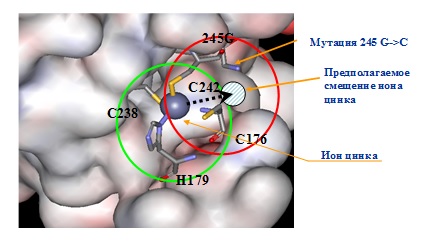
8) С помощью программы предсказания функциональных сайтов в пространственных структур белков PDBSiteScan и методов молекулярной динамики изучен структурный эффект действия мутации Gly245-Сys в ДНК-связывающем домене белка p53, ассоциированной с наследственной предрасположенностью к раку (синдром Ли-Фраумени). Показано, что мутация приводит к появлению нового сайта связывания иона цинка в непосредственной близости с существующим цинк-связывающим сайтом (рис. 8). Смещение иона цинка в данный сайт ведет к существенным конформационным перестройкам белка, что может обуславливать нарушения его функций (Pintus et al, 2013).
Рисунок 8. Графическое изображение фрагмента пространственной структуры ДНК-связывающего домена белка p53, в котором согласно расчетам в результате мутации Gly245-Сys возникает новый цинк-связывающий сайт.
5. Задачи, планируемые на перспективу: формулировки должны содержать как фундаментальные научные задачи, так и возможную прикладную направленность исследований в подразделении. Белки представляют собой важнейшую компоненту ГС. Наряду со структурными, каталитическими, транспортными функциями, белки в ГС выполняют важнейшие регуляторные функции: контролируют экспрессию генов, обеспечивают формирование сетей белок-белковых, РНК/ДНК-белковых и белок-лигандных регуляторных взаимодействий, путей передачи сигналов от клеточных мембран к ядрам клеток, межклеточных коммуникаций. Функциональная активность белков в ГС определяется наборами характерных для них функциональных сайтов. По данным базы PDB (Rose et al., 2011) в расшифрованных пространственных структурах белков может содержаться от нескольких до десяти и более функциональных сайтов. Однако, база данных PDB содержит в настоящее время описание 3-D структур только для ~ 80 000 белков. Из них уникальные пространственные структуры составляют ~ 15%, а остальные являются изоформами или мутантными формами. При этом в PDB представлено около 20 000 уникальных 3-D структур белков человека, имеющих гомологию менее 95%. Таким образом, до сих пор имеется большой разрыв между информацией, представленной в базе данных и полным количеством белков, кодируемых геномом человека с учетом вариантов альтернативного сплайсинга, что затрудняет оценку влияния генетической изменчивости на функциональные сайты белков. Поэтому важнейшее значение имеет создание высокопроизводительных конвейерных методов компьютерного предсказания функциональных сайтов в пространственных структурах белков. В связи с этим планируется решение следующих задач из области компьютерного анализа структурно-функциональной организации белков, функционирующих в биомедицински значимых ГС человека: предсказание компьютерными методами функциональных сайтов в пространственных структурах этих белков; оценка на этой основе репертуаров белок-белковых, РНК/ДНК-белковых и метаболит-белковых взаимодействий, и реконструкция сетей межмолекулярных взаимодействий, значимых для функционирования изучаемых ГС; картирование аминокислотных полиморфизмов на функциональные сайты белков и изучение на этой основе влияния мутаций на структуру и функцию белков, а также на формируемые ими сети межмолекулярных взаимодействий; изучение влияния мутаций на термодинамическую стабильность и специфическую активность белков, а также на структурные и конформационные свойства функциональных сайтов в трехмерной структуре белков. Короткие природные пептиды обладают широкой биологической активностью, включая регуляцию деятельности высшей нервной системы. Механизмы их действия до сих пор остаются слабо изученными. Наша идея состоит в том, что пептиды могут взаимодействовать с рецепторами за счет структурной мимикрии функциональных сайтов белков, для которых эти рецепторы являются мишенями. Будет создана база данных выравниваний последовательностей коротких пептидов с конформационными пептидами белков, характеризующая потенциальные молекулярные функции коротких пептидов и их мишени по аналогии с белками, имеющими сходство с этими пептидами. Будет создан параллельный алгоритм для предсказания функциональных сайтов в 3-D структурах белков на основе метода PDBSiteScan. Будут предсказаны функциональные сайты в пространственных структурах белков, функционирующих в составе изучаемых ГС, включая сайты, возникающие в результате генетической изменчивости, с помощью новой версии программы. Особое внимание будет обращено на функциональные сайты белков, выполняющих регуляторные функции (сайты аллостерической регуляции белков; сайты связывания РНК/ДНК, сайты, участвующее в формировании сетей белок-белковых взаимодействий). Будет создана реляционная версия базы данных PDBSite, интегрирующая информацию по экспериментально установленным и предсказанным (потенциальным) сайтам в 3-D структуре белков. Будет проведена классификация сайтов по сходству их пространственных структур, физико-химических и конформационных свойств, создание обобщенных шаблонов, описывающих различные структурные и функциональные классы сайтов. Будет осуществлена интеграция PDBSite с внешними базами данных Gene Ontology (GO), UniProt, PDB и др. Будет осуществлено расширение базы данных PDBSite: картирование микроэволюционной (популяционной) и макроэволюционной генетической изменчивости на пространственные структуры и функциональные сайты белков, функционирующих в составе изучаемых ГС, включая: - популяционные полиморфизмы из баз данных "1000 геномов", dbSNP, OMIM и др. - нуклеотидные и аминокислотные замены, выявляемые во множественных выравниваниях семейств белков, функционирующих в составе изучаемых ГС; - особое внимание будет обращено на аминокислотные замены в белках, выполняющих в исследуемых ГС регуляторные функции, а также ассоциированных с социально значимыми заболеваниями человека. Будет осуществлено развитие методов молекулярного моделирования динамики и механики белков для анализа функциональных сайтов и их взаимодействия с различными лигандами, включая малые химические соединения, белки, молекулы РНК и регуляторные сайты в ДНК. Будет разработан параллельный алгоритм для оценки изменений термодинамической стабильности мутантных белков на основе методов ProtStability. Будет исследовано влияние аминокислотных замен, представленных в базе данных PDBSite, на термодинамическую стабильность и специфическую активность белков, в том числе белков, функционирующих в составе изучаемых ГС. Будет исследовано влияние аминокислотных замен, представленных в базе данных PDBSite, на структурные и конформационные свойства функциональных сайтов белков, включая сайты, локализованные в белках, функционирующих в составе изучаемых ГС. Будет изучено влияние замен аминокислот на конформационную подвижность сайтов, изменения их физико-химических характеристик, устойчивость сетей водородных связей, формируемых аминокислотными остатками функциональных сайтов и т.д. Будет создан параллельный алгоритм для предсказания величин специфических активностей белков на основе разработанного нами ранее метода WebProAnalyst, использующего в качестве исходной информации множественные выравнивания семейств белков с известными величинами их специфической активности. Будет проведен анализ влияния аминокислотных замен, описанных в базе PDBSite, на специфическую активность белков, включая белки, функционирующие в составе изучаемых ГС. Будет проведена интеграция разработанных информационных и программных продуктов в систему «КОМПЬЮТЕРНАЯ ПРОТЕОМИКА» для конвейерного высокопроизводительного анализа структурно-функциональной организации белков и их функциональных сайтов, обеспечивающего: - предсказание пространственной (3-D) структуры белков по их аминокислотным последовательностям; - быстрый поиск структурной гомологии белков по базе данных PDB; - предсказание функциональных сайтов в 3-D структурах белков; - количественный анализ взаимосвязи «структура-активность» в семействах родственных и мутантных белков; - предсказание изменения термодинамической стабильности мутантных белков; - молекулярное моделирование динамики и механики белков; - оценку влияния аминокислотных замен в белках на их структурно-функциональные характеристики. Будет создана база знаний по вывяленным особенностям структурно-функциональной организации белков, функционирующих в составе изучаемых ГС, включая влияние мутаций на их структуру и функцию, и расширение на этой основе ГС. Антропова Евгения Александровна [младший научный сотрудник] Деменков Павел Сергеевич [научный сотрудник] Иванисенко Тимофей Владимирович [ведущий инженер] Мищенко Елена Леонидовна [научный сотрудник] Твердохлеб Наталья Николаевна [программист 1 категории] Яцык Иван Викторович [инженер-исследователь] Бывшие сотрудникиАлемасов Николай АлександровичАнарбаев Рашид Октамович Жамбалдагбаев Нима Цыренович Завьялов Владимир Юрьевич Иванисенко Никита Владимирович Медведева Ирина Вадимовна Петровский Евгений Дмитриевич Сайк Ольга Владимировна Степаненко Ирина Лембитовна СовместителиВолянская Анастасия Рашидовна [младший научный сотрудник]Выберите слайдером нужный промежуток, и список ниже будет содержать записи только нужного периода: 93 94 95 96 97 98 99 00 01 02 03 04 05 06 07 08 09 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 Публикации Монографии Конференции Гранты Научное руководство Учебные курсы Патенты
|
| 2025 | Pharmacological targeting of caspase-8/c-FLIPL heterodimer enhances complex II assembly and elimination of pancreatic cancer cells Corinna König, Nikita V Ivanisenko, Vladimir A Ivanisenko, Dagmar Kulms, Inna N Lavrik Commun. biolog., 2025 |
| Metabolomic and gene networks approaches reveal the role of mitochondrial membrane proteins in response of human melanoma cells to THz radiation Ekaterina A Butikova, Nikita V Basov, Artem D Rogachev, Evgeniy V Gaisler, Vladimir A Ivanisenko, Pavel S Demenkov, Aelita-Luiza A Makarova, Timofey V Ivanisenko, Ivan A Razumov, Daria A Kolomeyets, Sergey V Cheresiz, Olga I Solovieva, Kirill P Larionov, Yulia S Sotnikova, Yuri V Patrushev, Nikolay A Kolchanov, Andrey G Pokrovsky, Nikolay A Vinokurov, Vladimir V Kanygin, Vasiliy M Popik, Oleg A Shevchenko BBA-MOL CELL BIOL L, 2025 |
|
| 2024 | Кандидатные SNP-маркеры изменения экспрессии гена SCN9A человека в качестве интегратора генерации, чувства, ответа на боль и анестезии Доценко П.А., Золотарева К.А., Иванов Р.А., Чадаева И.В., Подколодный Н.Л., Иванисенко В.А., Деменков П.С., Лашин С.А., Пономаренко М.П. Vavilov journal of genetics and breeding, 2024, 8, 28, 808-821 |
| AthRiboNC: an Arabidopsis database for ncRNAs with coding potential revealed from ribosome profiling. Shen Y., Liu L., Liu E., Li S., Orlov Y., Ivanisenko V., Chen M. DATABASE-OXFORD, 2024, 2024 Dec 17;2024:baae123. doi: 10.1093/database/baae123. PMID: 39689042. |
|
| Multiple Myeloma: Genetic and Epigenetic Biomarkers with Clinical Potential Yuliya A. Veryaskina, Sergei E. Titov, Natalia V. Skvortsova, Igor B. Kovynev, Oksana V. Antonenko, Sergei A. Demakov, Pavel S. Demenkov, Tatiana I. Pospelova, Mikhail K. Ivanov, Igor F. Zhimulev IJMSCI, 2024, 25(24), 13404 |
|
| Could SLC26A7 Be a Promising Marker for Preoperative Diagnosis of High-Grade Papillary Thyroid Carcinoma? Sergei E. Titov,Evgeniya S. Kozorezova,Sergei A. Lukyanov ,Sergei V. Sergiyko ,Pavel S. Demenkov ,Yulia A. Veryaskina ,Sergey L. Vorobyev ,Ilya V. Sleptsov ,Roman A. Chernikov ,Natalia I. Timofeeva ,Svetlana V. Barashkova ,Elena L. Lushnikova ,Anna A. Uspenskaya ,Anna V. Zolotoukho ,Olga V. Romanova ,Igor F. Zhimulev Diagnostics, 2024, 14(23), 2652 |
|
| AI-Assisted Identification of Primary and Secondary Metabolomic Markers for Postoperative Delirium Ivanisenko VA, Rogachev AD, Makarova A-LA, Basov NV, Gaisler EV, Kuzmicheva IN, Demenkov PS, Venzel AS, Ivanisenko TV, Antropova EA, Kolchanov NA, Plesko VV, Moroz GB, Lomivorotov VV, Pokrovsky AG INT J MOL SCI, 2024, 25(21), 11847; |
|
| An Accurate and Efficient Approach to Knowledge Extraction from Scientific Publications Using Structured Ontology Models, Graph Neural Networks, and Large Language Models Ivanisenko TV, Demenkov PS, Ivanisenko VA INT J MOL SCI, 2024, 25(21), 11811; |
|
| MicroRNA: A Signature for the Clinical Progression of Chronic Lymphocytic Leukemia Yuliya A. Veryaskina,Sergei E. Titov,Igor B. Kovynev ,Tatiana I. Pospelova,Sofya S. Fyodorova,Yana Yu. Shebunyaeva ,Sergei A. Demakov ,Pavel S. Demenkov, Igor F. Zhimulev Lymphatics, 2024, 2024, 2(3), 157-167 |
|
| AtSNP_TATAdb: Candidate molecular markers of plant advantages related to single nucleotide polymorphisms within proximal promoters of Arabidopsis thaliana L. Bogomolov A, Zolotareva K, Filonov S, Chadaeva I, Rasskazov D, Sharypova E, Podkolodnyy N, Ponomarenko P, Savinkova L, Tverdokhleb N, Khandaev B, Kondratyuk E, Podkolodnaya O, Zemlyanskaya E, Kolchanov NA, Ponomarenko M. INT J MOL SCI, 2024, 1, 25, 607 |
|
| Modulation of extrinsic apoptotic pathway by intracellular glycosylation Kamil Seyrek, Nikita V. Ivanisenko, Corinna König, Inna N. Lavrik TRENDS CELL BIOL, 2024 |
|
| Prediction of the Aggressive Clinical Course of Papillary Thyroid Carcinoma Based on Fine Needle Aspiration Biopsy Molecular Testing Sergei A. Lukyanov, Sergei E. Titov, Evgeniya S. Kozorezova, Pavel S. Demenkov, Yulia A. Veryaskina, Denis V. Korotovskii, Tatyana E. Ilyina, Sergey L. Vorobyev, Vladimir A. Zhivotov, Nikita S. Bondarev, Ilya V. Sleptsov, Sergei V. Sergiyko IJMSCI, 2024, 25(13), 7090 |
|
| Design of Ctenophore Ca2+-Regulated Photoprotein Berovin Capable of Being Converted into Active Protein Under Physiological Conditions: Computational and
Experimental Approaches Ludmila P. Burakova, Nikita V. Ivanisenko, Natalia V. Rukosueva, Vladimir A. Ivanisenko, Eugene S. Vysotski Life, 2024 |
|
| Non-coding RNA notations, regulations and interactive resources Mengwei Cheng, Yinhuan Zhu, Han Yu, Linlin Shao, Yiming Zhang, Lanxing Li, Haohong Tu, Luyao Xie, Haoyu Chao, Peijing Zhang, Saige Xin, Cong Feng, Vladimir Ivanisenko, Yuriy Orlov, Dijun Chen, Aloysius Wong, Yixin Eric Yang & Ming Chen FUNCT INTEGR GENOMIC, 2024 |
|
| Candidate SNP markers significantly altering the affinity of the TATA-binding protein for the promoters of human genes associated with primary open-angle glaucoma Zolotareva K, Dotsenko P, Podkolodnyy N, Ivanov R, Makarova A-L, Chadaeva I, Bogomolov A, Demenkov P, Ivanisenko V, Oshchepkov D, Ponomarenko M. INT J MOL SCI, 2024, 23, 25, 12802 |
|
| Targeting type I DED interactions at the DED filament serves as a sensitive switch for cell fate decisions Corinna König, Nikita V. Ivanisenko, Laura K. Hillert-Richter, Deepti Namjoshi, Kalyani Natu, Johannes Espe, Dirk Reinhold, Nikolai A. Kolchanov, Vladimir A. Ivanisenko, Thilo Kähne, Kakoli Bose, Inna N. Lavrik Cell Chemical Biology, 2024 |
|
| Цифровая платформа «Микробиотех»: архитектура и назначение. Деменков П. С., Мухин А. М., Иванисенко В. А., Лашин С. А., Колчанов Н. А. Проблемы информатики, 2024 |
|
| Реконструкция и компьютерный анализ структурно-функциональной организации генной сети регуляции биосинтеза холестерина у человека и эволюционная характеристика участвующих в ней генов. Михайлова А.Д., Лашин С.А., Иванисенко В.А., Деменков П.С., Игнатьева Е.В. Vavilov journal of genetics and breeding, 2024, 28(8):864-873 |
|
| Онтологии в моделировании и анализе больших генетических данных Подколодный Н.Л., Подколодная О.А., Иванисенко В.А., Марченко М.А. Vavilov journal of genetics and breeding, 2024, 28(8):940-949 |
|
| Reconstruction and computer analysis of the structural and functional organization of the gene network regulating cholesterol biosynthesis in humans and the evolutionary characteristics of the genes involved in the network. Mikhailova A.D., Lashin S.A., Ivanisenko V.A., Demenkov P.S., Ignatieva E.V. Vavilov journal of genetics and breeding, 2024, 28(8):864-873. |
|
| Поиск перспективных генетических маркеров, ассоциированных с молекулярными механизмами снижения устойчивости риса к Rhizoctonia solani при избытке азотных удобрений, методом реконструкции и анализа генных сетей Е. А. Антропова, А. Р. Волянская, А. В. Адамовская, П. С. Деменков, И. В. Яцык, Т. В. Иванисенко, Ю. Л. Орлов, Х. Чао, М. Чэнь, В. А. Иванисенко Vavilov journal of genetics and breeding, 2024, 28(8):960-973 |
|
| Реконструкция и компьютерный анализ генной сети, отражающей роль микроРНК в регуляции ответа пшеницы на засуху М. А. Клещев, А. В. Мальцева, Е. А. Антропова, П. С. Деменков, Т. В. Иванисенко, Ю. Л. Орлов, Х. Чао, М. Чэнь, Н. А. Колчанов, В. А. Иванисенко Vavilov journal of genetics and breeding, 2024 |
|
| Изучение особенностей метаболизма тканей глиобластомы и перитуморального пространства при использовании таргетированного метаболомного скрининга методом ВЭЖХ-МС/МС и генных сетей Н. В. Басов, А. В. Адамовская, А. Д. Рогачев, Е. В. Гайслер, П. С. Деменков, Т. В. Иванисенко, А. С. Вензель, С. В. Мишинов, В. В. Ступак, С. В. Чересиз, О. С. Олешко, Е. А. Бутикова, А. Е. Осечкова, Ю. С. Сотникова, Ю. В. Патрушев, А. С. Поздняков, И. Н. Лаврик, В. А. Иванисенко, А. Г. Покровский Vavilov journal of genetics and breeding, 2024 |
|
| AthRiboNC: an Arabidopsis database for ncRNAs with coding potential revealed from ribosome profiling Shen Y, Liu L, Liu E, Li S, Orlov Y, Ivanisenko V, Chen M DATABASE-OXFORD, 2024, 2024 Dec 17;2024:baae123 |
|
| ПРОГРАММНЫЙ КОНВЕЙЕР ПРЕДСКАЗАНИЯ ВЛИЯНИЯ МУТАЦИЙ НА СТАБИЛЬНОСТЬ ПРОСТРАНСТВЕННЫХ СТРУКТУР БЕЛКОВ С ИСПОЛЬЗОВАНИЕМ МЕТОДОВ ОЦЕНКИ ИЗМЕНЕНИЯ СВОБОДНОЙ ЭНЕРГИИ И ИСКУССТВЕННОГО ИНТЕЛЛЕКТА Вензель Артур Сергеевич, Иванисенко Тимофей Владимирович, Деменков Павел Сергеевич, Иванисенко Владимир Александрович Проблемы информатики, 2024 |
|
| МЕТОД ПРЕДСКАЗАНИЯ КОЛИЧЕСТВА БЕЛКА В КЛЕТКАХ ДРОЖЖЕЙ НА ОСНОВЕ ИХ ГЕНОМНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ Вензель Артур Сергеевич, Клименко Александра Игоревна, Иванисенко Тимофей Владимирович, Деменков Павел Сергеевич, Лашин Сергей Александрович, Иванисенко Владимир Александрович Проблемы информатики, 2024 |
|
| 2023 | Differentially expressed genes and molecular susceptibility to human age-related diseases Shikhevich S, Chadaeva I, Khandaev B, Kozhemyakina R, Zolotareva K, Kazachek A, Oshchepkov D, Bogomolov A, Klimova NV, Ivanisenko VA, Demenkov P, Mustafin Z, Markel A, Savinkova L, Kolchanov NA, Kozlov V, Ponomarenko M INT J MOL SCI, 2023, 4, 24, 3996 |
| Candidate SNP markers significantly altering the affinity of TATA-binding protein for the promoters of human hub genes for atherogenesis, atherosclerosis and atheroprotection. Bogomolov A, Filonov S, Chadaeva I, Rasskazov D, Khandaev B, Zolotareva K, Kazachek A, Oshchepkov D, Ivanisenko VA, Demenkov P, Podkolodnyy N, Kondratyuk E, Ponomarenko P, Podkolodnaya O, Mustafin Z, Savinkova L, Kolchanov N, Tverdokhleb N, Ponomarenko M. INT J MOL SCI, 2023, 10, 24, 9010 |
|
| Первичная и вторичная микро-РНК модуляция внешнего пути апоптоза при гепатоцеллюлярной карциноме Хлебодарова Т.М., Деменков П.С., Иванисенко Т.В., Антропова Е.А., Лаврик И.Н., Иванисенко В.А. Молекулярная биология, 2023, 57(2):166–177 |
|
| Применение генных сетей к анализу результатов
метаболомного скрининга плазмы крови пациентов
с послеоперационным делирием В.А. Иванисенко, Н.В. Басов, А.А. Макарова, А.С. Вензель, А.Д. Рогачев, П.С. Деменков, Т.В. Иванисенко, М.А. Клещев, Е.В. Гайслер, Г.Б. Мороз, В.В. Плеско, Ю.С. Сотникова, Ю.В. Патрушев, В.В. Ломиворотов, А.Г. Покровский Vavilov journal of genetics and breeding, 2023, 27(7):768-775 |
|
| Молекулярно-генетические пути регуляции вирусом гепатита С экспрессии клеточных факторов PREB и PLA2G4C, играющих важную роль для репликации вируса Е.Л. Мищенко, А.А. Макарова, Е.А. Антропова, А.С. Вензель, Т.В. Иванисенко, П.С. Деменков, В.А. Иванисенко Vavilov journal of genetics and breeding, 2023, 27(7):776-783 |
|
| Accurate noise-robust classification of
Bacillus species from MALDI-TOF MS spectra
using a denoising autoencoder Yulia E. Uvarova, Pavel S. Demenkov, Irina N. Kuzmicheva, Artur S. Venzel, Elena L. Mischenko, Timofey V. Ivanisenko, Vadim M. Efimov, Svetlana V. Bannikova, Asya R. Vasilieva, Vladimir A. Ivanisenko and Sergey E. Peltek J INTEGR BIOINFORM, 2023 |
|
| Reconstruction of the regulatory hypermethylation network controlling hepatocellular carcinoma development during hepatitis C viral infection Evgeniya A. Antropova, Tamara M. Khlebodarova, Pavel S. Demenkov, Anastasiia R. Volianskaia, Artur S. Venzel, Nikita V. Ivanisenko, Alexandr D. Gavrilenko, Timofey V. Ivanisenko, Anna V. Adamovskaya, Polina M. Revva, Nikolay A. Kolchanov, Inna N. Lavrik and Vladimir A. Ivanisenko J INTEGR BIOINFORM, 2023 |
|
| Human_SNP_TATAdb – база данных о SNP, статистически достоверно изменяющих сродство ТАТА-связывающего белка к промоторам генов человека: полногеномный анализ и варианты использования. Филонов С.В., Подколодный Н.Л., Подколодная О.А., Твердохлеб Н.Н., Пономаренко П.М., Рассказов Д.А., Богомолов А.Г., Пономаренко М.П. Vavilov journal of genetics and breeding, 2023, 7, 27, 728-736 |
|
| Приоритизация потенциальных фармакологических мишеней для создания лекарств против гепатокарциномы, модулирующих внешний путь апоптоза, на основе реконструкции и анализа ассоциативных генных сетей П. С. Деменков, Е. А. Антропова, А. В. Адамовская, Е. Л. Мищенко, Т. М. Хлебодарова, Т. В. Иванисенко, Н. В. Иванисенко, А. С. Вензель, И. Н. Лаврик, В. А. Иванисенко Vavilov journal of genetics and breeding, 2023 |
|
| Реконструкция и анализ регуляторной генной сети функционирования клеточной стенки листьев Arabidopsis thaliana L. при ответе на водный дефицит A.R. Volyanskaya, E.A. Antropova, U.S. Zubairova, P.S. Demenkov, A.S. Venzel, Y.L. Orlov, A.A. Makarova, T.V. Ivanisenko, T.A. Gorshkova, A.R. Aglyamova, N.A. Kolchanov, M. Chen, V.A. Ivanisenko Vavilov journal of genetics and breeding, 2023 |
|
| Primary and Secondary micro-RNA Modulation the Extrinsic Pathway of Apoptosis in Hepatocellular Carcinoma Khlebodarova TM, Demenkov PS, Ivanisenko TV, Antropova EA, Lavrik IN, Ivanisenko VA. Molecular Biology, 2023, 57(2):165-175 |
|
| Prioritization of potential pharmacological targets for thedevelopment of anti-hepatocarcinoma drugs modulating the extrinsic apoptosis pathway: the reconstruction andanalysis of associative gene networks help. Demenkov P.S., Antropova E.A., Adamovskaya A.V., Mishchenko E.L., Khlebodarova T.M., Ivanisenko T.V., Ivanisenko N.V., Venzel A.S., Lavrik I.N., Ivanisenko V.A. Vavilov journal of genetics and breeding, 2023, 27(7):784-793 |
|
| PROSTATA: a framework for protein stability assessment
using transformers Dmitriy Umerenkov, Fedor Nikolaev, Tatiana I. Shashkova, Pavel V. Strashnov, Maria Sindeeva, Andrey Shevtsov, Nikita V. Ivanisenko, Olga L. Kardymon BIOINFORMATICS, 2023 |
|
| Apoptosis Genes as a Key to Identification of Inverse Comorbidity of Huntington's Disease and Cancer Bragina EY, Gomboeva DE, Saik OV, Ivanisenko VA, Freidin MB, Nazarenko MS, Puzyrev VP. INT J MOL SCI, 2023, May 27;24(11):9385 |
|
| Gene networks for use in metabolomic data analysis of blood plasma from patients with postoperative delirium Ivanisenko V.A., Basov N.V., Makarova A.A., Venzel A.S., Rogachev A.D., Demenkov P.S., Ivanisenko T.V., Kleshchev M.A., Gaisler E.V., Moroz G.B., Plesko V.V., Sotnikova Y.S., Patrushev Y.V., Lomivorotov V.V., Kolchanov N.A., Pokrovsky A.G. Vavilov journal of genetics and breeding, 2023, 27(7):768-775 |
|
| Integrating omics databases for enhanced crop breeding Chao H, Zhang S, Hu Y, Ni Q, Xin S, Zhao L, Ivanisenko VA, Orlov YL, Chen M J INTEGR BIOINFORM, 2023, Jul 25;20(4) |
|
| 2022 | Computer analysis of the relation between hydrogen bond stability in SOD1 mutants and the survival time of amyotrophic lateral sclerosis patients Alemasov, N. A., Timofeev, V. S., Ivanisenko, N. V., Kolchanov, N. A., & Ivanisenko, V. A. J MOL GRAPH MODEL, 2022 |
| Impact of human CD95 mutations on cell death and autoimmunity: a model. Seyrek K., Ivanisenko N. V., Wohlfromm F., Espe J., Lavrik I. N. TRENDS IMMUNOL, 2022 |
|
| Regulation of extrinsic apoptotic signaling by c-FLIP: towards targeting cancer networks Nikita V. Ivanisenko, Kamil Seyrek, Laura K. Hillert Richter, Corinna König, Johannes Espe, Kakoli Bose, Inna N. Lavrik Trends in Cancer, 2022 |
|
| Plant_SNP_TATA_Z-tester: a Web service that unequivocally estimates the impact of proximal promoter mutations on plant gene expression Rasskazov D, Chadaeva I, Sharypova E, Zolotareva K, Khandaev B, Ponomarenko P, Podkolodnyy N, Tverdokhleb N, Vishnevsky O, Bogomolov A, Podkolodnaya O, Savinkova L, Zemlyanskaya E, Golubyatnikov V, Kolchanov N, Ponomarenko M. INT J MOL SCI, 2022, V. 23. N. 15. Article number 8684. |
|
| Plasma metabolomics and gene regulatory networks analysis reveal the role of nonstructural SARS-CoV-2 viral proteins in metabolic dysregulation in COVID-19 patients V. A. Ivanisenko, E. V. Gaisler, N. V. Basov, A. D. Rogachev, S. V. Cheresiz, T. V. Ivanisenko, P. S. Demenkov, E. L. Mishchenko, O. P. Khripko, Yu. I. Khripko, S. M. Voevoda, T. N. Karpenko, A. J. Velichko, M. I. Voevoda, N. A. Kolchanov, A. G. Pokrovsky SCI REP-UK, 2022, 12, 19977 |
|
| The New Version of the ANDDigest Tool with Improved AI-Based Short Names Recognition Timofey V. Ivanisenko, Pavel S. Demenkov, Nikolay A. Kolchanov, Vladimir A. Ivanisenko INT J MOL SCI, 2022, 23(23), 14934 |
|
| Компьютерный анализ особенностей регуляции гиперметилированных маркерных генов гепатокарциномы вирусными белками гепатита С. Антропова Е.А., Хлебодарова Т.М., Деменков П.С., Вензель А.С., Иванисенко Н.В., Гавриленко А.Д., Иванисенко Т.В., Адамовская А.В., Ревва П.М., Лаврик И.Н., Иванисенко В.А. Vavilov journal of genetics and breeding, 2022, 26(8):733-742 |
|
| Computer analysis of regulation of hepatocarcinoma marker genes hypermethylated by HCV proteins. Antropova E.A., Khlebodarova T.M., Demenkov P.S., Venzel A.S., Ivanisenko N.V., Gavrilenko A.D., Ivanisenko T.V., Adamovskaya A.V., Revva P.M., Lavrik I.N., Ivanisenko V.A. Vavilov journal of genetics and breeding, 2022, 26(8):733-742 |
|
| Sputum Microbiome Composition in Patients with Squamous Cell Lung Carcinoma E. D. Baranova, V. G. Druzhinin, L. V. Matskova, P. S. Demenkov, V. P . Volobaev, V. I. Minina, A. V. Larionov, V. A. Titov Life, 2022 |
|
| Sputum Microbiota in Coal Workers Diagnosed with Pneumoconiosis as Revealed by 16S rRNA Gene Sequencing Vladimir G. Druzhinin, Elizaveta D. Baranova, Ludmila V. Matskova, Pavel S. Demenkov, Valentin P. Volobaev, Varvara I. Minina, Alexey V. Larionov and Snezana A. Paradnikova Life, 2022 |
|
| Comparison of Sputum and Oropharyngeal Microbiome Compositions in Patients with Non-Small Cell Lung Cancer Elizaveta Baranova, Vladimir Druzhinin, Ludmila Matskova, Pavel Demenkov, Valentin Volobaev, Alexey Larionov OBM Genetics, 2022 |
|
| Репликационно-транскрипционный комплекс коронавирусов: функции индивидуальных вирусных неструктурных субъединиц, свойства и архитектура их комплексов Е.Л. Мищенко., В.А. Иванисенко Vavilov journal of genetics and breeding, 2022, 26(2):121-127 |
|
| Development of Small Molecules Targeting Procaspase-8 at the DISC J. Espe, N. V. Ivanisenko, L. K. Hillert-Richter, V. A. Ivanisenko, I. N. Lavrik Cell and Tissue Biology, 2022 |
|
| Plant Biology and Biotechnology: Focus on Genomics and Bioinformatics Orlov Y.L., Ivanisenko V.A., Dobrovolskaya O.B., Chen M. INT J MOL SCI, 2022, 23, 6759 |
|
| 2021 | Hypoglycemia, Vascular Disease and Cognitive Dysfunction in Diabetes: Insights from Text Mining-Based Reconstruction and Bioinformatics Analysis of the Gene Networks Saik O.V., Klimontov V.V. INT J MOL SCI, 2021, Vol. 22. P. 12419. |
| Preoperative Typing of Thyroid and Parathyroid Tumors with a Combined Molecular Classifier. Titov SE, Kozorezova ES, Demenkov PS, Veryaskina YA, Kuznetsova IV, Vorobyev SL, Chernikov RA, Sleptsov IV, Timofeeva NI, Ivanov MK. Cancers, 2021, 13(2):237 |
|
| Correlation of Metabolic Profiles of Plasma and Cerebrospinal Fluid of High-Grade Glioma Patients A. D. Rogachev, N. A. Alemasov, V. A. Ivanisenko, N. V. Ivanisenko, E. V. Gaisler, O. S. Oleshko, S. V. Cheresiz, S. V. Mishinov, V. V. Stupak, A. G. Pokrovsky Metabolites, 2021 |
|
| The Web Platform for Storing Biotechnologically Significant Properties of Bacterial Strains Aleksey M. Mukhin, Fedor V. Kazantsev, Alexandra I. Klimenko, Tatiana N. Lakhova, Pavel S. Demenkov, Sergey A. Lashin Lect Notes Comput Sci, 2021 |
|
| Механочувствительные молекулярные взаимодействия в атерогенных районах артерий: развитие атеросклероза Мищенко Е.Л., Мищенко А.М., Иванисенко В.А. Vavilov journal of genetics and breeding, 2021, 25(5), 552-561 |
|
| Prioritization of biological processes based on the reconstruction and analysis of associative gene networks describing the response of plants to adverse environmental factors P.S. Demenkov, Е.А. Oshchepkova, T.V. Ivanisenko, V.A. Ivanisenko Vavilov journal of genetics and breeding, 2021, 2021 Sep; 25(5): 580–592. |
|
| 384-P: Text Mining and Bioinformatic Identification of Genes Linking Glycemic Variability and Impaired Angiogenesis in Diabetes Saik O.V., Klimontov V. DIABETES, 2021, Vol. 70, Suppl. 1. P. 384-P |
|
| Искусственный интеллект в диабетологии Климонтов В.В., Бериков В.Б., Сайк О.В. Сахарный диабет, 2021, Т. 24, №2. С. 156-166 |
|
| Glucose Variability: How Does It Work? Klimontov V.V., Saik O.V., Korbut A.I. INT J MOL SCI, 2021, Vol. 22. P. 7783 |
|
| Mechanosensitive molecular interactions in atherogenic regions of the arteries: development of atherosclerosis. Mishchenko EL, Mishchenko AM, Ivanisenko VA Vavilov journal of genetics and breeding, 2021, Sep;25(5):552-561 |
|
| Genetic damage in lymphocytes of lung cancer patients is correlated to the composition of the respiratory tract microbiome. Druzhinin VG, Matskova LV, Demenkov PS, Baranova ED, Volobaev VP, Minina VI, Larionov AV, Titov VA, Fucic A. MUTAGENESIS, 2021 |
|
| Sputum Microbiome Composition in Patients With Squamous Cell Lung Carcinoma E. D. Baranova, V. G. Druzhinin, L. V. Matskova, P. S. Demenkov, V. P . Volobaev, V. I. Minina, A. V. Larionov, V. A. Titov SCI REP-UK, 2021 |
|
| 2020 | Design, Synthesis and Molecular Modeling Study of Conjugates of ADP and Morpholino Nucleosides as A Novel Class of Inhibitors of PARP-1, PARP-2 and PARP-3 Yuliya V. Sherstyuk, Nikita V. Ivanisenko, Alexandra L. Zakharenko, Maria V. Sukhanova, Roman Y. Peshkov, Ilia V. Eltsov , Mikhail M. Kutuzov, Tatiana A. Kurgina, Ekaterina A. Belousova, Vladimir A. Ivanisenko, Olga I. Lavrik, Vladimir N. Silnikov and Tatyana V. Abramova INT J MOL SCI, 2020 |
| Identification of Antimicrobial Peptides from Novel Lactobacillus fermentum Strain. Anna S. Pavlova, Georgii D. Ozhegov, Georgij P. Arapidi, Ivan O. Butenko, Eduard S. Fomin, Nikolai A. Alemasov, Dmitry A. Afonnikov, Dina R. Yarullina, Vadim T. Ivanov, Vadim M. Govorun, Airat R. Kayumov PROTEIN J, 2020, 39, p. 73–84 |
|
| Dissecting DISC regulation via pharmacological targeting of caspase-8/c-FLIPL heterodimer Laura K. Hillert, Nikita V. Ivanisenko, Denise Busse, Johannes Espe, Corinna König, Sergey E. Peltek, Nikolai A. Kolchanov, Vladimir A. Ivanisenko, Inna N. Lavrik CELL DEATH DIFFER, 2020 |
|
| Learning the changes of barnase mutants thermostability from structural fluctuations obtained using anisotropic network modeling Nikolay A.Alemasov, Nikita V. Ivanisenko, Vladimir A.Ivanisenko J MOL GRAPH MODEL, 2020, Volume 97, June 2020, 107572 |
|
| Отражение особенностей физиологической регуляции сердечного ритма в протеоме мочи практически здоровых молодых мужчин В. Б. Русанов, Л. Х. Пастушкова, А. Г. Гончарова, А. Г. Черникова, А. М. Носовский, О. В. Сайк, Д. Н. Каширина, А. Г. Бржзовский, А. С. Кононихин, А. Г. Любишева, И. М. Ларина Физиология человека, 2020, ФИЗИОЛОГИЯ ЧЕЛОВЕКА, 2020, том 46, № 2, с. 84–93 |
|
| Controlling Cell Death through Post-translational Modifications of DED Proteins Kamil Seyrek, Nikita V. Ivanisenko, Max Richter, Laura K. Hillert, Corinna König, Inna N. Lavrik TRENDS CELL BIOL, 2020 |
|
| Reflection of Heart Rate Physiological Regulation Parameters in the Urinary Proteome in Healthy Young Males V. B. Rusanov, L. H. Pastushkova, A. G. Goncharova, A. G. Chernikova, A. M. Nosovsky, O. V. Saik, D. N. Kashirina, A. G. Brzhozovskiy, A. S. Kononikhin, A. G. Lubisheva, I. M. Larina Human Physiology, 2020, ISSN 0362-1197, Human Physiology, 2020, Vol. 46, No. 2, pp. 182–190. © Pleiades Publishing, Inc., 2020. Russian Text © The Author(s), 2020, published in Fiziologiya Cheloveka, 2020, Vol. 46, No. 2, pp. 84–93. |
|
| Taxonomic diversity of sputum microbiome in lung cancer patients and its relationship with chromosomal aberrations in blood lymphocytes V. G. Druzhinin, L. V. Matskova, P. S. Demenkov, E. D. Baranova, V. P. Volobaev, V. I. Minina, S. V. Apalko, M. A. Churina, S. A. Romanyuk, S. G. Shcherbak, V. I. Ivanov, A. V. Larionov SCI REP-UK, 2020 |
|
| ANDDigest: a new web-based module of ANDSystem for the search of knowledge in the scientific literature Timofey V. Ivanisenko, Olga V. Saik, Pavel S. Demenkov, Nikita V. Ivanisenko, Alexander N. Savostianov, Vladimir A. Ivanisenko BMC BIOINFORMATICS, 2020, 21, 228 (2020) |
|
| The role of death domain proteins in host response upon SARS-CoV-2 infection: modulation of programmed cell death and translational applications Nikita V. Ivanisenko, Kamil Seyrek, Nikolay A. Kolchanov, Vladimir A. Ivanisenko, Inna N. Lavrik Cell Death Discovery, 2020 |
|
| Mathematical Modeling Reveals the Importance of the DED Filament Composition in the Effects of Small Molecules Targeting Caspase-8/c-FLIPL Heterodimer N. V. Ivanisenko, I. N. Lavrik BIOCHEMISTRY-MOSCOW+, 2020 |
|
| Young «oil site» of the Uzon Caldera as a habitat for unique microbial life Sergey E. Peltek, Alla V. Bryanskaya, Yuliya E. Uvarova, Aleksey S. Rozanov, Timofey V. Ivanisenko, Vladimir A. Ivanisenko, Elena V. Lazareva, Olga V. Saik, Vadim M. Efimov, Sergey M. Zhmodik, Oxana P. Taran, Nikolay M. Slynko, Sergey V. Shekhovtsov, Valentin N. Parmon, Nikolay L. Dobretsov, Nikolay A. Kolchanov BMC MICROBIOL, 2020, 20, 349 |
|
| Pharmacological targeting of c-FLIPL and Bcl-2 family members promotes apoptosis in CD95L-resistant cells. Corinna König, Laura K. Hillert-Richter, Nikita V. Ivanisenko, Vladimir A. Ivanisenko, Inna N. Lavrik SCI REP-UK, 2020 |
|
| Preoperative detection of malignancy in fine-needle aspiration cytology (FNAC) smears with indeterminate cytology (Bethesda III, IV) by a combined molecular classifier Sergei Titov, Pavel S Demenkov, Sergei A Lukyanov, Sergei V Sergiyko, Gevork A Katanyan, Yulia A Veryaskina, Mikhail K Ivanov J CLIN PATHOL, 2020, J Clin Pathol 73(11): 722-727, 2020 |
|
| Bioinformatic reconstruction and analysis of gene networks related to glucose variability in diabetes and its complications Olga V Saik, Vadim V Klimontov INT J MOL SCI, 2020, 18;21(22):8691 |
|
| Состав бактериального микробиома в мокроте больных раком легкого и оценка его влияния на кластогенные эффекты в лимфоцитах крови. Дружинин В.Г., Баранова Е.Д., Волобаев В.П., Деменков П.С., Мацкова Л.В. Медицинская генетика, 2020, 19(9):98-100. |
|
| The story of the lost twins: decoding the genetic identities of the Kumhar and Kurcha populations from the Indian subcontinent. Das R, Ivanisenko VA, Anashkina AA, Upadhyai P. BMC GENET, 2020, Oct 22;21(Suppl 1):117 |
|
| 2019 | Improved regression model to predict an impact of SOD1 mutations on ALS patients survival time based on analysis of hydrogen bond stability Nikolay A.Alemasov, Nikita V. Ivanisenko, Bhupesh Taneja, Vibha Taneja, Srinivasan Ramachandran, Vladimir A. Ivanisenko J MOL GRAPH MODEL, 2019, Volume 86, January 2019, Pages 247-255 |
| A new version of the ANDSystem tool for automatic extraction of knowledge from scientific publications with expanded functionality for reconstruction of associative gene networks by considering tissue-specific gene expression Vladimir A. Ivanisenko, Pavel S. Demenkov, Timofey V. Ivanisenko, Elena L. Mishchenko, Olga V. Saik BMC BIOINFORMATICS, 2019, BMC Bioinformatics (2019) 20(Suppl 1): 34 |
|
| DNase and RNase activities of fresh cow milk lactoferrin Svetlana E. Soboleva, Olga D. Zakharova, Sergey E. Sedykh, Nikita V. Ivanisenko, Valentina N. Buneva, Georgy A. Nevinsky J MOL RECOGNIT, 2019 |
|
| ESEEM Reveals Bound Substrate Histidine in the ABC Transporter HisQMP2 Nikolay Isaev, Johanna Heuveling, Nikita Ivanisenko, Erwin Schneider, Heinz‑Jürgen Steinhof APPL MAGN RESON, 2019 |
|
| Prioritization of genes involved in endothelial cell apoptosis by their implication in lymphedema using an analysis of associative gene networks with ANDSystem Olga V. Saik, Vadim V. Nimaev, Dilovarkhuja B. Usmonov, Pavel S. Demenkov, Timofey V. Ivanisenko, Inna N. Lavrik, Vladimir A. Ivanisenko BMC MED GENOMICS, 2019, BMC Medical Genomics//2019;12(Suppl 2);47:117-140 |
|
| Organization and evolution of the chalcone synthase gene family in bread wheat and relative species Anastasia Y. Glagoleva, Nikita V. Ivanisenko, Elena K. Khlestkina BMC GENET, 2019, 20 (Suppl 1) :30 |
|
| Delineating the role of c-FLIP/NEMO interaction in the CD95 network via rational design of molecular probes Nikita V. Ivanisenko, Jörn H. Buchbinder, Johannes Espe, Max Richter, Miriam Bollmann, Laura K. Hillert, Vladimir A. Ivanisenko, Inna N. Lavrik BMC GENOMICS, 2019 |
|
| Приоритизация генов картофеля, вовлеченных в формирование селекционно-значимых признаков, с использованием базы знаний SOLANUM TUBEROSUM. Деменков П.С., Сайк О.В., Иванисенко Т.В., Колчанов Н.А., Кочетов А.В., Иванисенко В.А. Vavilov journal of genetics and breeding, 2019, 23(3):312-319 |
|
| Поиск белков протеома крови-регуляторов костного ремоделирования у космонавтов Л. Х. Пастушкова, А. Г. Гончарова, Г. Ю. Васильева, С. К. Тагирова, Д. Н. Каширина, О. В. Сайк, Й. Риттвегер, И. М. Ларина Физиология человека, 2019, ФИЗИОЛОГИЯ ЧЕЛОВЕКА, 2019, том 45, № 5, с. 91–98 |
|
| Search for Blood Proteome Proteins Involved in the Regulation of Bone Remodeling in Astronauts L. Kh. Pastushkovaa, A. G. Goncharovaa, G. Yu. Vasilyevaa, S. K. Tagirovaa, D. N. Kashirinaa, O. V. Saik, J. Rittwegerd, I. M. Larina Human Physiology, 2019, ISSN 0362-1197, Human Physiology, 2019, Vol. 45, No. 5, pp. 536–542. © Pleiades Publishing, Inc., 2019. Russian Text © The Author(s), 2019, published in Fiziologiya Cheloveka, 2019, Vol. 45, No. 5, pp. 91–98. |
|
| Mechanisms of Procaspase-8 Activation in the Extrinsic Programmed Cell Death Pathway Ivanisenko N.V., Lavrik I.N. Molecular Biology, 2019 |
|
| Long and short isoforms of c-FLIP act as control checkpoints of DED filament assembly. Hillert LK, Ivanisenko NV, Espe J, König C, Ivanisenko VA, Kähne T, Lavrik IN ONCOGENE, 2019 |
|
| Combined quantitation of HMGA2 mRNA, microRNAs, and mitochondrial-DNA content enables the identification and typing of thyroid tumors in fine-needle aspiration smears. Titov SE, Ivanov MK, Demenkov PS, Katanyan GA, Kozorezova ES, Malek AV, Veryaskina YuA, Zhimulev IF BMC CANCER, 2019 |
|
| Comorbidity of asthma and hypertension may be mediated by shared genetic dysregulation and drug side effects. Zolotareva O, Saik OV, Königs C, Bragina EY, Goncharova IA, Freidin MB, Dosenko VE, Ivanisenko VA, Hofestädt R. SCI REP-UK, 2019, Nov 8;9(1):16302 |
|
| Web-Based Computational Tools for the Prediction and Analysis of Posttranslational Modifications of Proteins Ivanisenko VA, Ivanisenko TV, Saik OV, Demenkov PS, Afonnikov DA, Kolchanov NA. Methods in Molecular Biology, 2019, 1934:1-20 |
|
| The molecular mechanisms driving physiological changes after long duration space flights revealed by quantitative analysis of human blood proteins. Kashirina DN, Percy AJ, Pastushkova LK, Borchers CH, Kireev KS, Ivanisenko VA, Kononikhin AS, Nikolaev EN, Larina IM. BMC MED GENOMICS, 2019, Mar 13;12(Suppl 2):45 |
|
| Evaluation of cardiovascular system state by urine proteome after manned space flight L Kh Pastushkova, DN Kashirina, AG Brzhozovskiy, AS Kononikhin, ES Tiys, VA Ivanisenko, MI Koloteva, EN Nikolaev, IM Larina ACTA ASTRONAUT, 2019, Том 160, стр. 594-600 |
|
| 2018 | How human serum albumin recognizes DNA and RNA. Alinovskaya L. I., Sedykh S. E., Ivanisenko N. V., Soboleva S. E., Nevinsky G. A. BIOL CHEM, 2018 |
| Molecular mechanisms underlying the impact of mutations in SOD1 on its conformational properties associated with amyotrophic lateral sclerosis as revealed with molecular modelling Nikolay A. Alemasov, Nikita V. Ivanisenko, Srinivasan Ramachandran, Vladimir A. Ivanisenko BMC STRUCT BIOL, 2018, Vol. 18 (Suppl 1), No. 1 |
|
| FunGeneNet: a web tool to estimate enrichment of functional interactions in experimental gene sets Tiys E.S., Ivanisenko T.V., Demenkov P.S., Ivanisenko V.A. BMC GENOMICS, 2018, 19, Suppl 3, p.103-116 |
|
| Novel candidate genes important for asthma and hypertension comorbidity revealed from associative gene networks Olga V. Saik, Pavel S. Demenkov, Timofey V. Ivanisenko, Elena Yu Bragina, Maxim B. Freidin, Irina A. Goncharova, Victor E. Dosenko, Olga I. Zolotareva, Ralf Hofestaedt, Inna N. Lavrik, Evgeny I. Rogaev, Vladimir A. Ivanisenko BMC MED GENOMICS, 2018, BMC Medical Genomics 2018, 11(Suppl 1)15:61-76 |
|
| Antibodies against H3 and H4 histones from the sera of HIV‐infected patients catalyze site‐specific degradation of these histones. Journal of Molecular Recognition. Baranova S. V., Dmitrenok P. S., Zubkova A. D., Ivanisenko N. V., Odintsova E. S., Buneva V. N., Nevinsky G. A. J MOL RECOGNIT, 2018 |
|
| Анализ взаимодействия генов нейронального апоптоза в ассоциативной генной сети болезни Паркинсона М.А. Янкина, О.В. Сайк, П.С. Деменков, Э.К. Хуснутдинова, Е.И. Рогаев, И.Н. Лаврик, В.А. Иванисенко Vavilov journal of genetics and breeding, 2018, Вавиловский журнал генетики и селекции. 2018;22(1):153-160 |
|
| База знаний SOLANUM TUBEROSUM: раздел по молекулярно-генетической регуляции метаболических путей Т.В. Иванисенко, О.В. Сайк, П.С. Деменков, В.К. Хлесткин, Е.К. Хлесткина, Н.А. Колчанов, В.А. Иванисенко Vavilov journal of genetics and breeding, 2018, Вавиловский журнал генетики и селекции. 2018;22(1):8-17 |
|
| Поиск микроРНК, потенциально задействованных в поддержании самообновления плюрипотентных клеток лабораторной крысы Шерстюк В.В., Медведев С.П., Ри М.Т., Вяткин Ю.В., Сайк О.В., Штокало Д.Н., Закиян С.М. Vavilov journal of genetics and breeding, 2018, Т. 22, № 2, С. 179-186 |
|
| Сytoscape – плагин для построения структурных моделей биологических сетей в виде случайных графов Подколодный Н.Л., Гаврилов Д.А., Твердохлеб Н.Н., Подколодная О.А. Вестник НГУ. Серия: Информационные технологии, 2018, номер 3, том 16, стр. 37-50. |
|
| Оценка методов приоритизации генов внешнего пути апоптоза в качестве кандидатов, ассоциированных с большим депрессивным расстройством М. А. Янкина, О. В. Сайк, В. А. Иванисенко, П. С. Деменков, Э. К. Хуснутдинова Генетика, 2018, Том 54, № 11, 1338-1348 |
|
| Ассоциативная генная сеть апоптоза и ее роль в механизмах коморбидности астмы и гипертонии Сайк О.В., Деменков П.С., Лаврик И.Н., Иванисенко В.А. Дневник науки, 2018, Дневник науки. 2018. №9. URL: http://www.dnevniknauki.ru/images/publications/2018/9/biology/Saik_Demenkov_Lavrik_Ivanisenko.pdf |
|
| Компьютерная база данных для анализа дифференциально экспрессирующихся генов, связанных с агрессивным поведением, на моделях лабораторных животных Анатолий Олегович Брагин, Кирилл Александрович Табанюхов, Ирина Витальевна Чадаева, Антон Витальевич Цуканов, Роман Олегович Бабенко, Ирина Вадимовна Медведева, Антон Геннадьевич Богомолов, Владимир Николаевич Бабенко, Юрий Львович Орлов Вестник НГУ. Серия: Информационные технологии, 2018, 2018. - Том 16, Выпуск № 3. - С. 7-21. - DOI 10.25205/1818-7900-2018-16-3-7-21. - ISSN 1818-7900 |
|
| ГЕНЫ ЭНДОТЕЛИАЛЬНОГО АПОПТОЗА КАК КАНДИДАТЫ ДЛЯ ИССЛЕДОВАНИЯ ИХ СВЯЗИ С ЛИМФЕДЕМОЙ О.В. Сайк, В.В. Нимаев, Д.Б. Усмонов, П.С. Деменков, И.Н. Лаврик, В.А. Иванисенко Международный журнал гуманитарных и естественных наук, 2018, Международный журнал гуманитарных и естественных наук. – 2018. – №. 10-1. - С. 22-27. |
|
| Компьютерное предсказание патогенных свойств мутантов белка SOD1, ассоциированных с боковым амиотрофическим склерозом Алемасов Н.А., Иваниесенко Н.В., Иванисенко В.А. Международный журнал гуманитарных и естественных наук, 2018, №10-1, С. 6-9 |
|
| АНАЛИЗ ОСОБЕННОСТЕЙ ГЕННОЙ СЕТИ ВНЕШНЕГО ПУТИ АПОПТОЗА ПРИ УЧЕТЕ ТКАНЕСПЕЦИФИЧНОЙ ЭКСПРЕССИИ ГЕНОВ С ПОМОЩЬЮ СИСТЕМЫ ANDSYSTEM Сайк О.В., Деменков П.С., Иванисенко В.А. Аллея науки, 2018, Аллея науки. – 2018. – №. 10(26) |
|
| Molecular Relationships between Bronchial Asthma and Hypertension as Comorbid Diseases Elena Yu. Bragina, Irina A. Goncharova, Anna F. Garaeva, Evgeniy V. Nemerov, Anastasija A. Babovskaya, Andrey B. Karpov, Yulia V. Semenova, Irina Z. Zhalsanova, Densema E. Gomboeva, Olga V. Saik, Olga I. Zolotareva, Vladimir A. Ivanisenko, Victor E. Dosenko, Ralf Hofestaedt, Maxim B. Freidin J INTEGR BIOINFORM, 2018, Journal of Integrative Bioinformatics. 2018; 20180052 |
|
| Search for New Candidate Genes Involved in the Comorbidity of Asthma and Hypertension Based on Automatic Analysis of Scientific Literature Olga V. Saik, Pavel S. Demenkov, Timofey V. Ivanisenko, Elena Yu., Maxim B. Freidin, Victor E. Dosenko, Olga I. Zolotareva, Evgeniy L. Choynzonov, Ralf Hofestaedt, Vladimir A. Ivanisenko J INTEGR BIOINFORM, 2018, Journal of Integrative Bioinformatics. 2018; Volume 15, Issue 4; 20180054 |
|
| shRNA-Induced Knockdown of a Bioinformatically Predicted Target IL10 Influences Functional Parameters in Spontaneously Hypertensive Rats with Asthma Drevytska T, Morhachov R, Tumanovska L, Portnichenko G, Nagibin V, Boldyriev O, Lapikova-Bryhinska T, Gurianova V, Dons'koi B, Freidin M, Ivanisenko V, Bragina EY, Hofestädt R, Dosenko V. J INTEGR BIOINFORM, 2018 |
|
| ПРОГРАММА ALLPRED ДЛЯ ПРЕДСКАЗАНИЯ АЛЛЕРГЕННОСТИ БАКТЕРИЙ И АРХЕЙ Брагин АО, Соколов ВС, Деменков ПС, Иванисенко ТВ, Брагина ЕЮ, Матушкин ЮГ, Иванисенко ВА Molecular Biology, 2018, №2, т. 52, с. 326-332 |
|
| Распределение сдвиговых напряжений на стенках артерий как главный фактор атерогенеза: численные и клинические исследования Мищенко Е.Л., Мищенко А.М., Иванисенко В.А. Дневник науки, 2018, №9, раздел Биологические науки |
|
| Выявление и анализ динамических паттернов суточной экспрессии генов млекопитающих О.А. Подколодная, Н.Н. Твердохлеб, Н.Л. Подколодный Vavilov journal of genetics and breeding, 2018, 8,22,1055-1062 |
|
| Prediction of Bacterial and Archaeal Allergenicity with AllPred Program Bragin A, Sokolov V, Demenkov P, Ivanisenko T, Bragina EY, Matushkin YG and Ivanisenko V Molecular Biology, 2018, Vol. 52(2), pp. 279-284 |
|
| Evaluation of Prioritization Methods of Extrinsic Apoptotic Signaling Pathway Genes for Retrieval of the New Candidates Associated with Major Depressive Disorder Yankina, M. A.; Saik, O. V.; Ivanisenko, V. A.; Demenkov, P. S. & Khusnutdinova, E. K. RUSS J GENET+, 2018, 54(11), 1366--1374. |
|
| Integrative Analysis of Co-Morbid Multifactorial Diseases. Hofestädt R, Ivanisenko V. J INTEGR BIOINFORM, 2018, Dec 15;15(4) |
|
| GenCoNet - A Graph Database for the Analysis of Comorbidities by Gene Networks. Shoshi A, Hofestädt R, Zolotareva O, Friedrichs M, Maier A, Ivanisenko VA, Dosenko VE, Bragina EY. J INTEGR BIOINFORM, 2018, Dec 25;15(4) |
|
| 2017 | SITEX 2.0: Projections of protein functional sites on eukaryotic genes. Extension with orthologous genes. Medvedeva IV, Demenkov PS, Ivanisenko VA Journal of Bioinformatics and Computational Biology, 2017, 1650044. doi: 10.1142/S021972001650044X. [Epub ahead of print] |
| РАЗРАБОТКА МЕТОДОВ АВТОМАТИЧЕСКОГО ИЗВЛЕЧЕНИЯ ЗНАНИЙ ИЗ ТЕКСТОВ НАУЧНЫХ ПУБЛИКАЦИЙ ДЛЯ СОЗДАНИЯ БАЗЫ ЗНАНИЙ SOLANUM TUBEROSUM О.В. САЙК, П.С. ДЕМЕНКОВ, Т.В. ИВАНИСЕНКО, Н.А. КОЛЧАНОВ, В.А. ИВАНИСЕНКО Сельскохозяйственная биология, 2017, 2017, том 52, №1, с. 63-74 |
|
| Computational modeling of the cell autonomous mammalian circadian oscillator Podkolodnaya O.A., Tverdokhleb N.N., Podkolodnyy N.L. BMC SYST BIOL, 2017, 11(Suppl 1):18 |
|
| Molecular mechanisms of the interaction between the processes of the cell response to mechanical stress and neuronal apoptosis in primary open-angle glaucoma O. V. Saik, N. A. Konovalova, P. S. Demenkov, N. V. Ivanisenko, T. V. Ivanisenko, D. E. Ivanoshchuk, O. S. Konovalova, O. A. Podkolodnaya, I. N. Lavrik, N. A. Kolchanov, V. A. Ivanisenko Russian Journal of Genetics: Applied Research, 2017, ISSN 2079-0597, 2017, Vol. 7, No. 5, pp. 558–564 |
|
| Regression model for predicting pathogenic properties of SOD1 mutants based on the analysis of conformational stability and conservation of hydrogen bonds. Alemasov N. A., Ivanisenko N. V., Ivanisenko V. A. J MOL GRAPH MODEL, 2017 |
|
| Компьютерные средства анализа транскриптомных данных: программный комплекс ExpGene А. М. Спицина, А. О. Брагин, А. И. Дергилев, И. В. Чадаева, Н. Н. Твердохлеб, Э. Р. Галиева, Л. Э. Табиханова, Ю. Л. Орлов Програмные системы: теория и приложения, 2017, 8:2(33), 45–68. |
|
| Приоритезация генов нейронального апоптоза по их структурной роли в ассоциативной генной сети нарушений аутического спектра с помощью подходов ANDsystem И.Н. Лаврик, В.А. Иванисенко, П.С. Деменков, О.В. Сайк, М.А. Янкина Международный журнал гуманитарных и естественных наук, 2017, №9, С: 5-11 |
|
| Анализ взаимодействия генов нейронального апоптоза и генов большого депрессивного расстройства в его ассоциативной генной сети Янкина М.А., Сайк О.В., Деменков П.С., Хуснутдинова Э.К., Лаврик И.Н., Иванисенко В.А. Дневник науки, 2017, №9'2017, С: 1-14 |
|
| Genome-wide profiling and differential expression of microRNA in rat pluripotent stem cells Sherstyuk V.V., Medvedev S.P., Elisaphenko E.A., Vaskova E.A., Ri M.T., Vyatkin Y.V., Saik O.V., Shtokalo D.N., Pokushalov E.A., Zakian S.M. SCI REP-UK, 2017, V. 7. - № 1. - P. 2787 |
|
| A study of structural properties of gene network graphs for mathematical modeling of integrated mosaic gene networks Olga V. Petrovskaya, Evgeny D. Petrovskiy, Inna N. Lavrik and Vladimir A. Ivanisenko Journal of Bioinformatics and Computational Biology, 2017 |
|
| Computer Analysis of Glioma Transcriptome Profiling: Alternative Splicing Events Babenko V.N., Gubanova N.V., Bragin A.O, Chadaeva I.V., Vasiliev G.V., Medvedeva I.V., Gaytan A.S., Krivoshapkin A.L., Orlov Yu.L. J INTEGR BIOINFORM, 2017, 20170022 DOI: 10.1515/jib-2017-0022 |
|
| АНАЛИЗ ВЛИЯНИЯ РАЗЛИЧНОГО УРОВНЯ СОЛЕПОТРЕБЛЕНИЯ НА БЕЛКОВЫЙ СОСТАВ МОЧИ В 105-СУТОЧНОЙ ИЗОЛЯЦИИ С ПОМОЩЬЮ ПРОГРАММЫ opoSOM Л. Х. Пастушкова, Д. Н. Каширина, А. Г. Бржозовский, В. А. Иванисенко, Е. С. Тийс, А. С. Кононихин, Н. Л. Стародубцева, Е. Н. Николаев, Г. Биндер, И. М. Ларина Физиология человека, 2017, том 43, № 1, с. 89–96 |
|
| ПОИСК ГЕНОВ-КАНДИДАТОВ, АССОЦИИРОВАННЫХ С ТРЕВОЖНЫМ НЕВРОЗОМ, НА ОСНОВЕ АВТОМАТИЧЕСКОГО АНАЛИЗА НАУЧНЫХ ТЕКСТОВ С ПОМОЩЬЮ ANDSYSTEM Янкина М.А.,Сайк О.В.,Деменков П.С.,Хуснутдинова Э.К.,Иванисенко В.А. Аллея науки, 2017, 2017. Т. 2. № 14. С. 712-720. |
|
| ИНТЕЛЛЕКТУАЛЬНЫЙ АНАЛИЗ ДАННЫХ В ПОИСКЕ НОВЫХ МИШЕНЕЙ ПАТОГЕНЕЗА ЛИМФЕДЕМЫ Усманов Д.Б. , Сайк О.В. , Нимаев В.В. Трансляционная медицина, 2017, Приложение № 3, с. 39 |
|
| Search of MicroRNAs Regulating the Receptor Status of Breast Cancer In Silico and Experimental Confirmation of Their Expression in Tumors V. S. Chernyi, P. V. Tarasova, V. V. Kozlov, O. V. Saik, N. E. Kushlinskii, L. F. Gulyaeva B EXP BIOL MED+, 2017, Vol. 163, No. 5, 2017 |
|
| Biodiversity of the microbial mat of the Garga hot spring Rozanov A. S., Bryanskaya A. V., Ivanisenko T. V., Malup T. K., Peltek S. E. BMC EVOL BIOL, 2017 |
|
| Integrated mathematical models for describing complex biological processes. Mishchenko E. L., Petrovskaya O. V., Mishchenko A. M., Petrovskiy E. D., Ivanisenko N. V., Ivanisenko V. A. BIOPHYSICS, 2017, 62(5), 778-795 |
|
| Роль генов апоптоза в контроле агрессивного поведения, выявленная с помощью комбинированного анализа ассоциативных генных сетей, экспрессионных и геномных данных по серым крысам с агрессивным поведением А.О. Брагин, О.В. Сайк, И.В. Чадаева, П.С. Деменков, А.Л. Маркель, Ю.Л. Орлов, Е.И. Рогаев, И.Н. Лаврик, В.А. Иванисенко Vavilov journal of genetics and breeding, 2017, 21(8):911-919 |
|
| A Metagenomic Study of Benthic Microbial Communities of Saline Lakes of the Novosibirsk Oblast A. V. Bryanskaya, Yu. E. Uvarova, A. S. Rozanov, T. K. Malup, E. V. Lazareva, O. P. Taran, O.P. Daguruva, Т. V. Ivanisenko, S. E.Peltek 13th International conference on salt lake research. Ulan-Ude, Buryat State University Publishing Department., 2017, P.76 |
|
| Анализ циркадного ритма биологических процессов в печени и почках мыши Подколодный Н.Л. Твердохлеб Н.Н. Подколодная О.А. Vavilov journal of genetics and breeding, 2017, Т. 21, N 8, с. 903-910 |
|
| 2016 | Dynamic properties of SOD1 mutants can predict survival time of patients carrying familial amyotrophic lateral sclerosis Nikolay A. Alemasova, Nikita V. Ivanisenko, Sergey P. Medvedev, Suren M. Zakian, Nikolay A. Kolchanov & Vladimir A. Ivanisenko Journal Of Biomolecular Structure & Dynamics, 2016, 2017, Volume 35, Issue 3, Pages 645-656 |
| Mosaic gene network modelling identified new regulatory mechanisms in HCV infection. Popik OV, Petrovskiy ED, Mishchenko EL, Lavrik IN, Ivanisenko VA. VIRUS RES, 2016, Oct 22. pii: S0168-1702(15)30083-6. doi: 10.1016/j.virusres.2015.10.004. |
|
| Functional annotation of the vlinc class of non-coding RNAs using systems biology approach. Laurent GS, Vyatkin Y, Antonets D, Ri M, Qi Y, Saik O, Shtokalo D, de Hoon MJ, Kawaji H, Itoh M, Lassmann T, Arner E, Forrest AR; FANTOM consortium, Nicolas E, McCaffrey TA, Carninci P, Hayashizaki Y, Wahlestedt C, Kapranov P. NUCLEIC ACIDS RES, 2016, Nucleic Acids Res. 2016 Mar 21. pii: gkw162 |
|
| Компьютерное моделирование пространственных структур пептидов из MUC1, способных ингибировать апоптоз Н.В. Иванисенко, И.Н. Лаврик, В.А. Иванисенко Vavilov journal of genetics and breeding, 2016, 19(6):731-737 |
|
| Дизайн и проверка действия малых химических соединений, направленных на ингибирование белка FADD Н.В. Иванисенко, Л. Хиллерт, В.А. Иванисенко, И.Н. Лаврик Vavilov journal of genetics and breeding, 2016, 2015;19(6):724-730 |
|
| NACE: A web-based tool for prediction of intercompartmental efficiency of human molecular genetic networks Olga V. Popik, Timofey V. Ivanisenko, Olga V. Saik, Evgeny D. Petrovskiy, Inna N. Lavrik, Vladimir A. Ivanisenko VIRUS RES, 2016, DOI information: 10.1016/j.virusres.2015.11.029 |
|
| Characteristics of Age-Dependent Changes in Urine Proteome in Healthy Men L. Kh. Pastushkova, A. S. Kononikhin, E. S. Tiys, I. V. Dobrokhotov, V. A. Ivanisenko, E. N. Nikolaev, I. M. Larina, I. A. Popov Adv. Gerontol, 2016, Vol. 6, No. 2, pp. 122–127 |
|
| Candidate SNP markers of chronopathologies are predicted by a significant change in the affinity of TATA-binding protein for human gene promoters Petr Ponomarenko, Dmitry Rasskazov, Valentin Suslov, Ekaterina Sharypova, Ludmila Savinkova, Olga Podkolodnaya, Nikolay Podkolodny, Natalya Tverdokhleb, Irina Chadaeva, Mikhail Ponomarenko, Nikolay Kolchanov Biomed Res Int, 2016, vol. 2016, Article ID 8642703. doi:10.1155/2016/8642703 |
|
| Interactome of the hepatitis C virus: Literature mining with ANDSystem Saik OV, Ivanisenko TV, Demenkov PS, Ivanisenko VA VIRUS RES, 2016, Jun 15;218:40-8 |
|
| Biomedical and candidate SNP markers of chronopathologies can significantly change the affinity of the ТАТА-binding protein to the promoters of human genes. Rasskazov DA, Podkolodnyy NL, Podkolodnaya OA, Tverdokhleb NN, Suslov VV, Savinkova LK, Ponomarenko PM, Ponomarenko MP Russian Journal of Genetics: Applied Research, 2016, 7, 6, 738-748 |
|
| Effects of SNPs in the positioning regions of RNA polymerase II on the TBP/promoter affinity in genes of the human circadian clock. Podkolodnaya OA, Rasskazov DA, Podkolodnyy NL, Podkolodnaya NN, Suslov VV, Savinkova LK, Ponomarenko PM, Ponomarenko MP. Russian Journal of Genetics: Applied Research, 2016, 7, 6, 759-770 |
|
| Молекулярно-генетические механизмы регуляции процессов апоптоза белками вируса гепатита С. Сайк О. В., Деменков П. С., Иванисенко Н. В., Иванисенко Т. В., Лаврик И. Н., Иванисенко В. А. Проблемы современной науки и образования, 2016, № 27 (69), 6-14. |
|
| МИКРОБНЫЕ СООБЩЕСТВА ЭКСТРЕМАЛЬНЫХ ЭКОСИСТЕМ; МЕТАГЕНОМНЫЙ ПОДХОД С.Е. Пельтек, А.В. Брянская, Ю.Е. Уварова, А.С. Розанов, Т.В. Иванисенко, Т.К. Малуп, В.А. Иванисенко, Е.В. Лазарева, О.В. Сайк, С.М. Жмодик, О.П. Таран, Н.М. Слынько, С.В. Шеховцов, В.Н. Пармон, Н.Л. Добрецов, Н.А. Колчанов Acta Naturae, 2016 |
|
| Математическая модель циркадного осциллятора млекопитающих: взаимодействие с системой NAD+/SIRT1 и возрастные изменения экспрессии генов циркадного осциллятора Подколодный Н.Л., Твердохлеб Н.Н., Подколодная О.А. Vavilov journal of genetics and breeding, 2016, 20(6):848-856 |
|
| Молекулярно-генетические механизмы взаимодействия процессов ответа клетки на механический стресс и нейронального апоптоза при первичной открытоугольной глаукоме Сайк О.В., Коновалова Н.А., Деменков П.С., Иванисенко Н.В., Иванисенко Т.В., Иванощук Д.Е., Пономарева М.Н., Коновалова О.С., Подколодная О.А., Лаврик И.Н., Колчанов Н.А., Иванисенко В.А. Vavilov journal of genetics and breeding, 2016, 20(6):840-847 |
|
| Analysis of differential gene expression by RNA-seq data in brain areas of laboratory animals Babenko V.N., Bragin A.O., Spitsina A.M., Chadaeva I.V., Galieva E.R., Orlova G.V., Medvedeva I.V., Orlov Y.L. J INTEGR BIOINFORM, 2016, 13(4):292. doi:10.2390/biecoll-jib-2016-292 |
|
| A compendium of human genes regulating feeding behavior and body weight, its functional characterization and identification of GWAS genes involved in brain-specific PPI network Elena V. Ignatieva, Dmitry A. Afonnikov, Olga V. Saik, Evgeny I. Rogaev and Nikolay A. Kolchanov BMC GENET, 2016, 17(Suppl 3):158 |
|
| Selection and validation of miRNAs as normalizers for profiling expression of microRNAs isolated from thyroid fine needle aspiration smears. Titov SE, Demenkov PS, Ivanov MK, Malakhina ES, Poloz TL, Tsivlikova EV, Ganzha MS, Shevchenko SP, Gulyaeva LF, Kolesnikov NN ONCOL REP, 2016, Oncol Rep. 2016 Nov;36(5):2501-2510. doi: 10.3892/or.2016.5113. Epub 2016 Sep 20. |
|
| Novel tuberculosis susceptibility candidate genes revealed by the reconstruction and analysis of associative networks Elena Yu. Bragina, Evgeny S. Tiys, Alexey A. Rudko, Vladimir A. Ivanisenko, Maxim B. Freidin Infection, Genetics and Evolution, 2016, Volume 46, December 2016, Pages 118–123 |
|
| Molecular associations of Primary Open-Angle Glaucoma with potential comorbid diseases (POAG-associome) Olga V Saik, Natalia A Konovalova, Pavel S Demenkov, Timofey V Ivanisenko, Evgeny D Petrovskiy, Nikita V Ivanisenko, Dinara E Ivanoshchuk, Maria N Ponomareva, Olga S Konovalova, Inna N. Lavrik, Nikolay A Kolchanov, Vladimir A Ivanisenko Biotecnología Aplicada, 2016, 2016;33:3201-3206 |
|
| Структурное моделирование мод связывания НАД+ с ПАРП-1 Иванисенко Н.В., Жечев Д.А., Иванисенко В.А. Vavilov journal of genetics and breeding, 2016 |
|
| Metabolic model of central carbon and energy metabolisms of growing Arabidopsis thaliana in relation to sucrose translocation Zakhartsev M., Medvedeva I., Orlov Y., Akberdin I., Krebs O., Schulze W.X. BMC PLANT BIOL, 2016, 2016 Vol.16:262 DOI: 10.1186/s12870-016-0868-3 |
|
| 2015 | Molecular association of pathogenetic contributors to pre-eclampsia (pre-eclampsia associome) Glotov AS, Tiys ES, Vashukova ES, Pakin VS, Demenkov PS, Saik OV, Ivanisenko TV, Arzhanova ON, Mozgovaya EV, Zainulina MS, Kolchanov NA, Baranov VS, Ivanisenko VA BMC SYST BIOL, 2015, 2015;9 Suppl 2:S4. doi: 10.1186/1752-0509-9-S2-S4 |
| Effect of flanking sequences on the accuracy of the recognition of transcription factor binding sites. Khlebodarova T. M., Oshchepkov D. Yu., Levitsky V. G., Podkolodnaya O. A., Ignatieva E. V., Ananko E. A., Stepanenko I. L., Kolchanov N. A. Russian Journal of Genetics: Applied Research, 2015, Vol. 5, No. 4, p. 322–329 |
|
| Structural and dynamic properties of mutant SOD1 proteins associated with amyotrophic lateral sclerosis Alemasov, N. A., Ivanisenko, N. V., Ivanisenko, V. A. Russian Journal of Genetics: Applied Research, 2015 |
|
| Identification of biological processes on the composition of the urine proteome in cosmonauts on the first day after long space flights Pastushkova LH, Kononihin AS, Tijs ES, Obraztsova OA, Dobrokhotov IV, Ivanisenko VA, Nikolaev EN, Larina IM Российский физиологический журнал имени И.М. Сеченова, 2015, 2015 Feb;101(2):222-37 |
|
| Permanent proteins in the urine of healthy humans during the Mars-500 experiment Larina IM, Pastushkova LKh, Tiys ES, Kireev KS, Kononikhin AS, Starodubtseva NL, Popov IA, Custaud MA, Dobrokhotov IV, Nikolaev EN, Kolchanov NA, Ivanisenko VA Journal of Bioinformatics and Computational Biology, 2015, 2015 Feb;13(1):1540001. doi: 10.1142/S0219720015400016. Epub 2015 Jan 8. |
|
| Kidney Function and Urine Protein Composition in Healthy Volunteers During Space Station Fitness Tests Fomina, Elena V.; Lisova, Natalia Iu.; Kireev, Kirill S.; Tiys, Evgeny S.; Kononikhin, Alexey S.; Larina, Irina M. Aerospace Medicine and Human Performance, 2015, Volume 86, Number 5, May 2015, pp. 472-476(5) |
|
| Суперкомпьютерный анализ геномных и транскриптомных данных, полученных с помощью технологий высокопроизводительного секвенирования ДНК Спицина А.М., Орлов Ю.Л., Подколодная Н.Н., Свичкарев А.В., Дергилев А.И., Чен М., Кучин Н. В., Черных И. Г., Глинский Б. М. Программные системы: теория и приложения, 2015, 6:1(23), c. 157–174 |
|
| Shifts in urine protein profile during dry immersion. Pastushkova L Kh, Kononikhin AS, Tiys ES, Nosovsky AM, Dobrokhotov IV, Ivanisenko VA, Nikolaev EN, Novoselova NM, Custaud MA, Larina IM. Aviakosm Ekolog Med., 2015, 2015;49(4):15-9. |
|
| Computer analysis of protein functional sites projection on exon structure of genes in Metazoa. Medvedeva I., Demenkov P., Ivanisenko V. BMC GENOMICS, 2015, 16(Suppl 13):S2. |
|
| Prediction of tissue-specific effects of gene knockout on apoptosis in different anatomical structures of human brain. Petrovskiy E.D., Saik O.V., Tiys E.S., Lavrik I.N., Kolchanov N.A., Ivanisenko V.A. BMC GENOMICS, 2015, 16(Suppl 13):S3 |
|
| The Mammalian Circadian Clock: Gene Regulatory Network and Computer Analysis O.A. Podkolodnaya, N.N. Podkolodnaya, N.L. Podkolodnyy Russian Journal of Genetics: Applied Research, 2015, Vol. 5, No. 4, pp. 354–362 |
|
| Биомедицинские и кандидатные SNP-маркеры для хронопатологий могут достоверно изменять сродство ТАТА-связывающего белка к промоторам генов человека. Рассказов Д.А., Подколодный Н.Л., Подколодная О.А., Подколодная Н.Н., Суслов В.В., Савинкова Л.К., Пономаренко П.М., Пономаренко М.П. Vavilov journal of genetics and breeding, 2015, 6, 19, 691-698 |
|
| Влияние однонуклеотидных полиморфных замен в районах позиционирования РНК-полимеразы II на сродство к ним TBP в генах циркадных часов человека Подколодная О.А., Рассказов Д.А., Подколодный Н.Л., Подколодная Н.Н., Суслов В.В., Савинкова Л.К., Пономаренко П.М., Пономаренко М.П. Vavilov journal of genetics and breeding, 2015, 6, 19, 682-690 |
|
| ВЫЯВЛЕНИЕ ЗНАЧИМО ПРЕДСТАВЛЕННЫХ БИОЛОГИЧЕСКИХ ПРОЦЕССОВ ПО СОСТАВУ ПРОТЕОМА МОЧИ КОСМОНАВТОВ НА ПЕРВЫЕ СУТКИ ПОСЛЕ ДЛИТЕЛЬНЫХ КОСМИЧЕСКИХ ПОЛЕТОВ ПАСТУШКОВА Л.Х., КОНОНИХИН А.С., ТИЙС Е.С., ОБРАЗЦОВА О.А., ДОБРОХОТОВ И.В., ИВАНИСЕНКО В.А., НИКОЛАЕВ Е.Н., ЛАРИНА И.М. Российский физиологический журнал имени И.М. Сеченова, 2015, Том: 101 Номер: 2 Страницы: 222-237 |
|
| НОВЫЕ ГЕНЫ-КАНДИДАТЫ ПОДВЕРЖЕННОСТИ ТУБЕРКУЛЕЗУ, УСТАНОВЛЕННЫЕ С ПОМОЩЬЮ ПОСТРОЕНИЯ И АНАЛИЗА АССОЦИАТИВНЫХ СЕТЕЙ БРАГИНА Е.Ю., РУДКО А.А., ТИЙС Е.С., ИВАНИСЕНКО В.А., ФРЕЙДИН М.Б. Бюллетень сибирской медицины, 2015, Том: 14Номер: 6 Год: 2015 Страницы: 33-39 |
|
| ANDSystem: an Associative Network Discovery System for automated literature mining in the field of biology Vladimir A Ivanisenko, Olga V Saik, Nikita V Ivanisenko, Evgeny S Tiys, Timofey V Ivanisenko, Pavel S Demenkov and Nikolay A Kolchanov BMC SYST BIOL, 2015, 9(Suppl 2):S2; doi:10.1186/1752-0509-9-S2-S2 |
|
| Interaction sorting method for molecular dynamics on multi-core SIMD CPU architecture Matvienko S., Alemasov N., and Fomin E. Journal of Bioinformatics and Computational Biology, 2015, Vol. 13. No 2. P. 1540004–1540018 |
|
| 2014 | The Proteome of a Healthy Human during Physical Activity under Extreme Conditions Larina I. M., Ivanisenko V. A., Nikolaev E. N., Grigorev A. I. Acta Naturae, 2014, VOL. 6 № 3 (22) 2014 |
| Предсказание генов маркеров агрессивности глиомы низкой степени злокачественности по экспрессионным данным TCGA Иванисенко Н.В., Губанова Н.В., Колчанов Н.А., Иванисенко В.А. Mathematical Biology & Bioinformatics, 2014, Т. 9, N 2, С. 534-542 |
|
| Bioinformatics analysis of the genome of Geobacillus stearothermophilus 22 Strain isolated from the Garga hot spring, Baikal Region Rozanov A.S., Ivanisenko T.V., Bryanskaya A.V., Shekhovtsov S.V., Logacheva M.D., Saik O.V., Malup T.K., Demenkov P.S., Goryachkovskaya T.N., Ivanisenko V.A., Peltek S.E. Russian Journal of Genetics: Applied Research, 2014 |
|
| Program complex SNP-MED for analysis of single-nucleotide polymorphism (SNP) effects on the function of genes associated with socially significant diseases N. L. Podkolodnyy, D. A. Afonnikov, Yu. Yu. Vaskin, L.O. Bryzgalov, V. A. Ivanisenko, P. S. Demenkov, M.P. Ponomarenko, D.A. Rasskazov, K. V. Gunbin, I. V. Protsyuk, I.Yu. Shutov, P.N. Leontyev, M.Yu. Fursov, N.P. Bondar, E.V. Antontseva, T.I. Merkulova, and N.A. Kolchanov Russian Journal of Genetics: Applied Research, 2014, Vol. 4, No. 3, pp. 159–167. |
|
| Структурные и динамические особенности мутантов белка SOD1, ассоциированных с боковым амиотрофическим склерозом Алемасов Н.А., Иванисенко Н.В., Иванисенко В.А. Vavilov journal of genetics and breeding, 2014, Т. 18. № 4/2. С. 920–927 |
|
| A New Stochastic Model for Subgenomic Hepatitis C Virus Replication Considers Drug Resistant Mutants Nikita V. Ivanisenko, Elena L. Mishchenko, Ilya R. Akberdin, Pavel S. Demenkov, Vitaly A. Likhoshvai, Konstantin N. Kozlov, Dmitry I. Todorov, Vitaly V. Gursky, Maria G. Samsonova, Alexander M. Samsonov, Diana Clausznitzer, Lars Kaderali, Nikolay A. Kolchanov, Vladimir A. Ivanisenko PloS One, 2014, Volume 9 | Issue 3 | e91502 |
|
| Exploring Interaction of TNF and Orthopoxviral CrmB Protein by Surface Plasmon Resonance and Free Energy Calculation. Ivanisenko NV, Tregubchak TV, Saik OV, Ivanisenko VA, Shchelkunov SN PROTEIN PEPTIDE LETT, 2014, Protein Pept Lett. 2014;21(12):1273-81. |
|
| Molecular dynamics simulations of the Nip7 proteins from the marine deep- and shallow-water Pyrococcus species Kirill E Medvedev, Nikolay A Alemasov, Yuri N Vorobjev, Elena V Boldyreva, Nikolay A Kolchanov, Dmitry A Afonnikov BMC STRUCT BIOL, 2014, 14:23 |
|
| Влияние фланкирующих последовательностей на точность распознавания сайтов связывания транскрипционных факторов. Хлебодарова Т.М., Ощепков Д.Ю., Левицкий В.Г., Подколодная О.А., Игнатьева Е.В., Ананько Е.А., Степаненко И.Л., Колчанов Н. А. Vavilov journal of genetics and breeding, 2014, т.18, № 4/2, с.876-886. |
|
| Molecular Analysis of the Benthos Microbial Community in Zavarzin Thermal Spring (Uzon Caldera, Kamchatka, Russia) Rozanov A.S., Bryanskaya A.V., Malup T.K., Lasareva E.V., Taran O.P., Ivanisenko T.V., Ivanisenko V.A., Zhmodik S.M., Kolchanov N.A., Peltek S.E. BMC GENOMICS, 2014 |
|
| An integrated study to analyze salt lake microbial community structure (Novosibirsk oblast, Russia) A. Bryanskaya, A. Rozanov, T. Malup, T. Aleshina, E. Lazareva, O. Taran, T. Goryachkovskaya, V. Ivanisenko, S. Peltek Acta Geologica Sinica (English Edition), 2014 |
|
| Insights into pathophysiology of dystropy through the analysis of gene networks: an example of bronchial asthma and tuberculosis Bragina Elena Yu, Evgeny S. Tiys, Maxim B. Freidin, Lada A. Koneva, Pavel S. Demenkov, Vladimir A. Ivanisenko, Nikolay A. Kolchanov, Valery P. Puzyrev IMMUNOGENETICS, 2014, no. 7-8 (2014): 457-465. |
|
| Identification of Proteins of Cardiovascular System in Healthy Subjects’ Urine during “Dry” Immersion L. Kh. Pastushkova, I. V. Dobrokhotov, O. M. Veselova, E. S. Tiys, A. S. Kononikhin, A. M. Novosiolova, M. Coupe, M.-A. Custaud, I. M. Larina Fiziol Cheloveka, 2014, Vol. 40, No. 3, pp. 330–339 |
|
| Постоянные белки мочи здорового человека в эксперименте с 520-суточной изоляцией Пастушкова ЛХ, Киреев КС, Кононихин АС, Тийс ЕС, Попов ИА, Доброхотов ИВ, Кусто МА, Иванисенко ВА, Колчанов НА, Николаев ЕН, Почуев ВИ, Ларина ИМ Aviakosm Ekolog Med., 2014, 48(1):48-54. |
|
| ЦИРКАДНЫЕ ЧАСЫ МЛЕКОПИТАЮЩИХ: ГЕННАЯ СЕТЬ И КОМПЬЮТЕРНЫЙ АНАЛИЗ Подколодная О.А., Подколодная Н.Н., Подколодный Н.Л. Vavilov journal of genetics and breeding, 2014, Т. 18. N. 4/2. стр. 928-938 |
|
| Analysis of signaling networks distributed over intracellular compartments based on protein-protein interactions Popik, O. V., Saik, O. V., Petrovskiy, E. D., Sommer, B., Hofestädt, R., Lavrik, I. N., Ivanisenko, V. A. BMC GENOMICS, 2014, 2014. – Т. 15. – №. Suppl 12. – С. S7 |
|
| Моделирование пространственного распределения эффекта нокаута генов, связанных с агрессивностью глиомы низкой степени злокачественности, в тканях мозга человека с помощью методов машинного обучения Петровский Е.Д., Колчанов Н.А., Иванисенко В.А. Mathematical Biology & Bioinformatics, 2014, Т. 9. № 2. С. 534-542. |
|
| 2013 | Computational screening for new inhibitors of M. tuberculosis mycolyltransferases antigen 85 group of proteins as potential drug targets. Gahoi S, Mandal RS, Ivanisenko N, Shrivastava P, Jain S, Singh AK, Raghunandanan MV, Kanchan S, Taneja B, Mandal C, Ivanisenko VA, Kumar A, Kumar R, Open Source Drug Discovery Consortium, Ramachandran S Journal Of Biomolecular Structure & Dynamics, 2013, 31(1):30-43 |
| The substitutions G245C and G245D in the Zn(2+)-binding pocket of the p53 protein result in differences of conformational flexibility of the DNA-binding domain. Pintus SS, Ivanisenko NV, Demenkov PS, Ivanisenko TV, Ramachandran S, Kolchanov NA, Ivanisenko VA. Journal Of Biomolecular Structure & Dynamics, 2013, 31:1, 78-86 |
|
| Subcellular localization charts: a new visual methodology for the semi-automatic localization of protein-related data sets. Sommer B., Kormeier B., Demenkov P., Arrigo P., Hippe K., Ates O., Kochetov A.V., Ivanisenko V., Kolchanov N.A., Hofestaedt R. Journal of Bioinformatics and Computational Biology, 2013, 11. 1. 1340005. |
|
| Биоинформатика: метод во главе угла Афонников Д.А. Иванисенко В.А. Наука из первых рук, 2013, 1,49,51-59 |
|
| Abundances of microRNAs in human cells can be estimated as a function of the abundances of YRHB and RHHK tetranucleotides in these microRNAs as an ill-posed inverse problem solution Ponomarenko MP, Suslov VV, Ponomarenko PM, Gunbin KV, Stepanenko IL, Vishnevsky OL, Kolchanov NA Frontiers in Genetics, 2013, , 4, 122 |
|
| PЕПЛИКАЦИЯ CУБГЕНОМНОГО PЕПЛИКОНА ВИPУCА ГЕПАТИТА C В ПPИCУТCТВИИ ИНГИБИТОPОВ NS3-ПPОТЕАЗЫ: CТОXАCТИЧЕCКАЯ МОДЕЛЬ Н.В. Иваниcенко, Е.Л. Мищенко, И.P. Акбеpдин, П.C. Деменков, В.А. Лиxошвай, К.Н. Козлов, Д.И. Тодоpов, М.Г. Cамcонова, А.М. Cамcонов, Н.А. Колчанов, В.А. Иваниcенко МОЛЕКУЛЯРНАЯ БИОФИЗИКА, 2013, БИОФИЗИКА, 2013, том 58, вып. 5, c. 758–774 |
|
| Accuracy of protein allergenicity prediction can be improved by taking into account data on allergenic protein discontinuous peptides. Bragin AO, Demenkov PS, Kolchanov NA, Ivanisenko VA. Journal Of Biomolecular Structure & Dynamics, 2013, 31(1):59-64. |
|
| Detection of renal and urinary tract proteins before and after spaceflight. Pastushkova LKh, Kireev KS, Kononikhin AS, Ivanisenko VA, Larina IM, Nikolaev EN AVIAT SPACE ENVIR MD, 2013, 84:859–863 |
|
| Detection of Renal Tissue and Urinary Tract Proteins in the Human Urine after Space Flight. Pastushkova L.Kh, Kireev R.S., Kononikhin A.S., Tiys E.S., Popov I.A., Starodubtseva N.L., Dobrokhotov I.V., Ivanisenko V.A., Larina I.M., Kolchanov N.A., Nikolaev E.N. PloS One, 2013, V8, e71652 |
|
| ОБНАРУЖЕНИЕ БЕЛКОВ ТКАНЕЙ ПОЧЕК И МОЧЕВЫВОДЯЩЕЙ СИСТЕМЫ В МОЧЕ ЧЕЛОВЕКА ПОСЛЕ КОСМИЧЕСКОГО ПОЛЕТА Л. Х. Пастушкова, К. С. Киреев, А. С. Кононихин, Е. С. Тийс, И. А. Попов, И. В. Доброхотов, В. А. Иванисенко, В. Б. Носков, И. М. Ларина, Е. Н. Николаев Физиология человека, 2013, том 39, № 5, с. 99–104 |
|
| Enhanced Differential Evolution Entirely Parallel Method for Biomedical Applications Konstantin Kozlov, Nikita Ivanisenko, Vladimir Ivanisenko, Nikolay Kolchanov, Maria Samsonova, Alexander M. Samsonov Lect Notes Comput Sci, 2013, Lecture Notes in Computer Science Volume 7979, 2013, pp 409-416 |
|
| ИЗУЧЕНИЕ ПРОТЕОМА МОЧИ ДЛЯ ОЦЕНКИ СОСТОЯНИЯ СЕРДЕЧНО-СОСУДИСТОЙ СИСТЕМЫ У ЧЕЛОВЕКА ПОСЛЕ КОСМИЧЕСКОГО ПОЛЕТА Пастушкова Л.Х., Кононихин А.С., Тийс Е.С., Попов И.А., Доброхотов И.В., Иванисенко В.А., Николаев Е.Н., Ларина И.М. Российский физиологический журнал имени И.М. Сеченова, 2013, Номер: 8, Том: 99, Страницы: 945-959 |
|
| Молекулярные механизмы взаимодействия белков CrmB вируса оспы коров и вируса натуральной оспы с фактором некроза опухолей человека Иванисенко Н.В., Трегубчак Т.В., Сайк О.В., Иванисенко В.А., Щелкунов С.Н., Колчанов Н.А. Mathematical Biology & Bioinformatics, 2013, Т. 8. № 2. С. 467–479. |
|
| Программный комплекс SNP-MED для анализа влияния однонуклеотидных полиморфизмов на функцию генов, связанных с развитием социально значимых заболеваний Н.Л. Подколодный, Д.А. Афонников, Ю.Ю. Васькин, Л.О. Брызгалов, В.А. Иванисенко, П.С. Деменков, М.П. Пономаренко, Д.А. Рассказов, К.В. Гунбин, И.В. Процук, И.Ю. Шутов, П.Н. Леонтьев, М.Ю. Фурсов, Н.П. Бондарь, Е.В. Антонцева, Т.И. Меркулова, Н.А. Колчанов. Vavilov journal of genetics and breeding, 2013, том 17, №4/1, с.577-588 |
|
| Биоинформатический анализ генома штамма Geobacilus stearothermophilus 22, выделенного из горячего источника Гарга (Прибайкалье). Розанов А.С., Иванисенко Т.В., Брянская А.В., Шеховцев С.В., Логачева М.Д., Сайк О.В., Малуп Т.К., Деменков П.С., Горячковская Т.Н., Иванисенко В.А., Пельтек С.Е. Vavilov journal of genetics and breeding, 2013, Т.17, С. 659-664 |
|
| Компьютерный анализ структуры липаз бактерий рода Geobacillus и выявление мотивов, влияющих на их термостабильность. Сорокина К.Н., Нуриддинов М.А., Розанов А.С., Иванисенко В.А., Пельтек С.Е. Vavilov journal of genetics and breeding, 2013, Т.17, С. 666-674 |
|
| 2012 | A Model of the Gene Network for Flowering Time Regulation in Winter Wheat and Barley. Smirnova O.G , Stepanenko I.L., Titov I.I. Russian Journal of Genetics: Applied Research, 2012, 2012, Vol. 2, No. 4, pp. 319–324 |
| Mechanism of Action and Activity Regulation of COP1, a Constitutive Repressor of Photomorphogenesis. Smirnova O.G , Stepanenko I.L., Shumny V.K. RUSS J PLANT PHYSL+, 2012, 2012, Vol. 59, No. 2, pp. 155–166. |
|
| SitEx: a computer system for analysis of projections of protein functional sites on eukaryotic genes Irina Medvedeva, Pavel Demenkov, Nikolay Kolchanov and Vladimir Ivanisenko NUCLEIC ACIDS RES, 2012, V. 40(Database issue):D278-83. |
|
| Механизм действия и регуляция активности конститутивного репрессора фотоморфогенеза COP1. Смирнова О.Г., Степаненко И.Л., Шумный В.К. Физиология растений, 2012, том 59, № 2, с. 1–13 |
|
| Protein Allergenicity Prediction on the Basis of Conformational Peptides A. O. Bragin, P. S. Demenkov, and V. A. Ivanisenko Russian Journal of Genetics: Applied Research, 2012, Russian Journal of Genetics: Applied Research, 2012, Vol. 2, No. 1, pp. 18–22. © Pleiades Publishing, Ltd., 2012 |
|
| Исследование термостабильности мутантных форм белка барназы с использованием программного комплекса MOLKERN Фомин Э.С., Алемасов Н.А. Vavilov journal of genetics and breeding, 2012, Т. 16, № 2, С. 415-426 |
|
| Компьютерный анализ метагеномных данных – предсказание количественной величины специфической активности белков Иванисенко В. А., Деменков П. С., Пинтус С. С., Иванисенко Т. В., Подколодный Н. Л., Иванисенко Л. Н., Розанов А. С., Брянская А. В., Кострюкова Е. С., Левицкий С. А., Селезнева О. В., Чукин М. М., Ларин А. К., Кондратов И. Г., Лазарев В. Н., Пельтек С. Е., Говорун В. М., Колчанов Н. А. Doklady Akademii Nauk, 2012, 443(2), 248-252 |
|
| Программный комплекс L-MOLKERN для расчетов разностей свободных энергий с учетом эффектов перераспределения заряда Фомин Э.С., Алемасов Н.А. Mathematical Biology & Bioinformatics, 2012, Т. 7. № 2. С. 398–409. URL: http://www.matbio.org/2012/Fomin_7_398.pdf |
|
| Модель генной сети регуляции времени цветения у озимой пшеницы и ячменя. Степаненко И.Л., Смирнова О.Г., Титов И.И. Vavilov journal of genetics and breeding, 2012, Т. 16, № 1, С. 99-108 |
|
| ICGenomics: программный комплекс анализа символьных последовательностей геномики Ю.Л. Орлов, А.О. Брагин, И.В. Медведева, К.В. Гунбин, П.С. Деменков, О.В. Вишневский, В.Г. Левицкий, Д.Ю. Ощепков, Н.Л. Подколодный, Д.А. Афонников, И. Гроссе, Н.А. Колчанов Vavilov journal of genetics and breeding, 2012, Том 16, 4/1, 732-741. |
|
| ПОДАВЛЕНИЕ РЕПЛИКАЦИИ СУБГЕНОМНОГО РНК РЕПЛИКОНА ВИРУСА ГЕПАТИТА С ИНГИБИТОРОМ NS3 ПРОТЕАЗЫ SCH 503034 В HUH-7 КЛЕТКАХ: СТОХАСТИЧЕСКАЯ МОДЕЛЬ Е.Л. Мищенко, Н.В. Иванисенко, И.Р. Акбердин, П.С. Деменков, В.А. Лихошвай, Н.А. Колчанов, В.А. Иванисенко Vavilov journal of genetics and breeding, 2012, 16(2), стр. 339-347 |
|
| Low-Temperature Dynamical and Structural Properties of Saturated and Monounsaturated Phospholipid Bilayers Revealed by Raman and Spin-Label EPR Spectroscopy. N. V. Surovtsev , N. V. Ivanisenko, K. Yu. Kirillov, S. A. Dzuba J PHYS CHEM B, 2012, 116 (28), pp 8139–8144 |
|
| Finding biomarkers in non-model species: literature mining of transcription factors involved in bovine embryo development. Turenne N, Tiys E, Ivanisenko V, Yudin N, Ignatieva E, Valour D, Degrelle S, Hue I BioData Mining, 2012, Aug 29;5(1):12 |
|
| Анализ белковых взаимодействий на основе изучения протеома мочи человека в эксперименте со 105-суточной изоляцией. Пастушкова Л.Х., Валеева О.А., Кононихин А.С., Ларина И.М., Николаев Е.Н., Попов И.А., Доброхотов И.В., Иванисенко В.А., Тийс Е.С., Колчанов Н.А. Авиакосмическая и экологическая медицина, 2012, Т. 46. № 2. С. 37-43 |
|
| Реконструкция ассоциативных белковых сетей, связанных с процессами регуляции обмена и депонирования натрия в организме здорового человека на основе изучения протеома мочи. Ларина И.М., Колчанов Н.А., Доброхотов И.В., Тийс Е.С., Деменков П.С., Иванисенко В.А., Валеева О.А., Пастушкова Л.Х., Николаев Е.Н. Физиология человека, 2012, т38, N3, 107-115 |
|
| Компьютерный анализ взаимосвязи аллергенности микроорганизмов и среды их обитания Брагин А.О., Деменков П.С., Тийс Е.С., Хофештадт Р., Иванисенко В.А. Vavilov journal of genetics and breeding, 2012, том 16 №4/1 784-790 |
|
| Распределенная система RESTful-web-сервисов для реконструкции и анализа генных сетей Подколодный Николай Леонтьевич Семенычев Александр Владимирович Рассказов Дмитрий Александрович Боровский Виктор Геннадьевич Ананько Елена Анатольевна Игнатьева Елена Васильевна Подколодная Наталья Николаевна Подколодная Ольга Александровна Колчанов Николай Александрович Vavilov journal of genetics and breeding, 2012, 4/1, Т.16, 791-798 |
|
| ANDVisio: a new tool for graphic visualization and analysis of literature mined associative gene networks in the ANDSystem P.S. Demenkov, T.V. Ivanisenko, N.A. Kolchanov, V.A. Ivanisenko In Silico Biology, 2012, 11(3), 149-161 |
|
| A Study of the Thermal Stability of Mutant Barnase Protein Variants with MOLKERN Software E.S. Fomin, N.A. Alemasov Russian Journal of Genetics: Applied Research, 2012, Vol. 2, No. 6, pp. 453–461 |
|
| Компьютерные методы исследования термостабильности белков и их применение в биологии Алемасов Н.А., Фомин Э.С. Vavilov journal of genetics and breeding, 2012, Вавиловский журнал генетики и селекции. 2012. Т. 16. № 4/1. С. 774–784. |
|
| Магнитно–резонансная спектроскопия метаболических изменений в мозге мышей при введении 2–дезокси–D–глюкозы и липополисахарида Мошкин М.П., Акулов А.Е., Петровский Д.В., Сайк О.В., Петровский Е.Д., Савелов А.А., Коптюг И.В. Российский физиологический журнал имени И.М. Сеченова, 2012, 98 (10): 1264-1273 |
|
| Computer analysis of metagenomic data--prediction of quantitative value of specific activity of proteins. Ivanisenko VA, Demenkov PS, Pintus SS, Ivanisenko TV, Podkolodny NL, Ivanisenko LN, Rozanov AS, Bryanskaya AV, Kostrjukova ES, Levizkiy SA, Selezneva OV, Chukin MM, Larin AK, Kondratov IG, Lazarev VN, Peltek SE, Govorun VM, Kolchanov NA Doklady Biochemistry and Biophysics, 2012, Mar-Apr;443:76-80. |
|
| Reconstruction of associative protein networks connected with processes of sodium exchange' regulation and sodium deposition in healthy volunteers by urine proteome analysis Larina IM, Kolchanov NA, Dobrokhotov IV, Ivanisenko VA, Demenkov PS, Tiĭs ES, Valeeva OA, Pastushkova LKh, Nikolaev EN Human Physiology, 2012, May-Jun;38(3):316-23 |
|
| 2011 | Protein Structure Discovery: пакет программ для решения задач компьютерной протеомики Иванисенко В. А., Деменков П. С., Иванисенко Т. В., Колчанов Н. А. Биоорганическая химия, 2011, 2011, Т. 37, № 1, С. 22–35 |
| Application of the ANDCell Computer System to Reconstruction and Analysis of Associative Networks Describing Potential Relationships between Myopia and Glaucoma O. A. Podkolodnaya, E. E. Yarkova, P. S. Demenkov, O. S. Konovalova, V. A. Ivanisenko, and N. A. Kolchanov Russian Journal of Genetics: Applied Research, 2011, 2011, Vol. 1, No. 1, pp. 21–28. |
|
| Извлечение знаний из текстов научных публикаций и создание баз знаний в области нанобиотехнологии В.А. Иванисенко, Н.Л. Подколодный, П.С. Деменков, Т.В. Иванисенко, О.А. Подколодная, Е.В. Игнатьева, Т.М. Хлебодарова, Н.Н. Подколодная, Е.А. Ананько, С.С. Гончаров, Н.А. Колчанов Российские нанотехнологии, 2011, 2011 . Т. 6 № 7- 8, с. 14-21. |
|
| PROMEDIA – база данных химических соединений, потенциальных биомаркеров заболеваний, имеющих значение для неинвазивной диагностики Сайк О.В., Мошкин М.П., Балдин М.Н., Грузнов В.М., Козлов В.А., Самороков С.Н., Деменков П.С., Иванисенко В.А., Колчанов Н.А. Mathematical Biology & Bioinformatics, 2011, №2, Т.6, С.250-263 |
|
| Предсказание аллергенности белков с использованием информации о конформационных пептидах А.О. Брагин, П.С. Деменков, В.А. Иванисенко Vavilov journal of genetics and breeding, 2011, 2011. Т. 15. № 3. С. 462-468. |
|
| The Role of the COP1, SPA, and PIF Proteins in Plant Photomorphogenesis O. G. Smirnova, I. L. Stepanenko, V. K. Shumnyia Biology Bulletin Reviews, 2011, Vol. 1, No. 4, pp. 314–324 |
|
| Роль белков COP1, SPA и PIF в регуляции фотоморфогенеза растений Смирнова О.Г., Степаненко И.Л., Шумный В.К. Успехи современной биологии, 2011, Т.311, 1. С.3-15 |
|
| Protein Structure Discovery: software package to perform computational proteomics tasks Ivanisenko VA, Demenkov PS, Ivanisenko TV, Kolchanov NA RUSS J BIOORG CHEM+, 2011, Jan-Feb;37(1):22-35. |
|
| 2010 | Использование компьютерной системы ANDCell для реконструкции и анализа ассоциативных сетей потенциальных механизмов взаимосвязи миопии и глаукомы О.А.Подколодная, Е.Э. Яркова, П.С.Деменков, О.С.Коновалова,. В.А.Иванисенко, Н.А.Колчанов Информационный вестник ВОГИС, 2010, Т. 14, № 1. — С. 106–115. |
| Характеристика C-концевой части гликопротеина Е вируса Западного Нила и оценка силы его взаимодействия с V 3 интегрином как с предполагаемым клеточным рецептором М. В. Богачек, Б. Н. Зайцев, S. K. Sekatskii, Е. В. Протопопова, В. А. Терновой, А. В. Иванова, А. В. Качко, В. А. Иванисенко, G. Dietler, В. Б. Локтев. BIOCHEMISTRY-MOSCOW+, 2010, том 75, вып. 4, с. 571 – 581 |
|
| Экспериментальная проверка функциональной активности потенциальных DRE-сайтов, обнаруженных в промоторах IRF1, REL и IL12A генов человека Е.В. Кашина, Е.В. Антонцева, М.Ю. Шаманина, Е.А. Ощепкова, Д.Ю. Ощепков, А.В. Катохин, А.Ю. Гришанова, Д.П. Фурман, В.А. Мордвинов Труды ТГУ. Серия: Биологическая, 2010, Том 275. С. 353-356. |
|
| Эффективная реализация алгоритма расчёта ближних невалентных взаимодействий на процессоре PowerXCell8i Алемасов Н.А., Фомин Э.С. Вычислительные технологии, 2010, №6, Т. 15, С. 3-18 |
|
| Expression and molecular characterization of the Mycobacterium tuberculosis PII protein Bandyopadhyay A, Arora A, Jain S, Laskar A, Mandal C, Ivanisenko VA, Fomin ES, Pintus SS, Kolchanov NA, Maiti S, Ramachandran S. J BIOCHEM, 2010, 147(2):279-89. |
|
| Functional Divergence of Helicobacter pylori Related to Early Gastric Cancer Momynaliev KT, Kashin SV, Chelysheva VV, Selezneva OV, Demina IA, Serebryakova MV, Alexeev D, Ivanisenko VA, Aman E, Govorun VM. J PROTEOME RES, 2010, 9(1):254-67 |
|
| Генная сеть активации макрофага: участие ксенобиотков в регуляции экспрессии генов цитокинов. Ощепкова Е.А., Ощепков Д.Ю., Кашина Е.В., Антонцева Е.В., Фурман Д.П., Мордвинов В.А. Цитокины и воспаление, 2010, Т.9, №3, с.49. |
|
| Visualization and analysis of a cardio vascular disease- and MUPP1-related biological network combining text mining and data warehouse approaches Sommer B, Tiys ES, Kormeier B, Hippe K, Janowski SJ, Ivanisenko TV, Bragin AO, Arrigo P, Demenkov PS, Kochetov AV, Ivanisenko VA, Kolchanov NA, Hofestädt R J INTEGR BIOINFORM, 2010, 7(1):148. doi: 10.2390/biecoll-jib-2010-148 |
|
| Characterization glycoprotein E С-end of West Nile virus and evaluation of its interaction force with αVβ3 integrin as putative cellular receptor Bogachek M.V., Zaitsev B. N., Sekatskii S. K., Protopopova E.V., Ternovoi V. A., Ivanova A.V., Kachko A.V., Ivanisenko V.A., Dietler G. Loktev V.B. BIOCHEMISTRY-MOSCOW+, 2010, 2010, Vol. 75, No. 4, pp. 472-480 |
|
| Веб-ориентированная система построения и исполнения комплексных запросов к реляционным источникам биологической информации на основе семантической формализации структуры данных Коротков Р.О., Деменков П.С., Иванисенко В.А. Вестник НГУ. Серия: Информационные технологии, 2010, Т. 8. № 3. С. 13-22. |
|
| База данных химических соединений, имеющих потенциальное значение для неинвазивной диагностики заболеваний Сайк О.В., Деменков П.С., Иванисенко Т.В., Мошкин М.П., Иванисенко В.А. В мире научных открытий, 2010, №4 (10), Часть 14. С. 102-103 |
|
| 2009 | Картирование рецепторного домена для вирусов Венесуэльского энцефалита лошадей и Клещевого энцефалита на С-конце ламининсвязывающего белка человека А.А. Малыгин, Е.И. Бондаренко, В.А. Иванисенко, Е.В. Протопопова, Г.Г. Карпова, В.Б. Локтев BIOCHEMISTRY-MOSCOW+, 2009, т. 74, вып. 12. с. 1631 – 1641 |
| Математическое моделирование действия потенциальных противовирусных препаратов на репликацию субгеномного репликона вируса гепатита С в клетке Е.Л. Мищенко, K.Д. Безматерных, В.А. Иванисенко, В.А. Лихошвай, Н.А. Колчанов Информационный вестник ВОГИС, 2009, №1, Том 13, 208-218 |
|
| Implementation of a Non-bonded Interaction Calculation Algorithm for the Cell Architecture Fomin E.S., Alemasov N.A. Lect Notes Comput Sci, 2009, Vol. 5698, P. 399–405 |
|
| Promoters of the Genes Encoding the Transcription Factors Regulating the Cytokine Gene Expression in Macrophages Contain Putative Binding Sites for Aryl Hydrocarbon Receptor D. P. Furman, E.A. Oshchepkova, D.Yu. Oshchepkov, M.Yu. Shamanina, V.A. Mordvinov COMPUT BIOL CHEM, 2009, V. 33. №6. P. 465-468. |
|
| Анализ распределения аденозин-фосфат связывающих сайтов белков на экзонной структуре гена Медведева И. В., Деменков П. С., Иванисенко В. А. Информационный вестник ВОГИС, 2009, Том 13, №1, стр. 122-127 |
|
| C-Terminal Fragment of Human Laminin-Binding Protein Contains a Receptor Domain for Venezuelan Equine Encephalitis and Tick-Borne Encephalitis Viruses Malygin A.A., Bondarenko E.I., Ivanisenko V.A., Protopopova E.V., Karpova G.G., Loktev V.B. BIOCHEMISTRY-MOSCOW+, 2009, Vol. 74, No. 12, pp. 1328 -1336 |
|
| Картирование рецепторного домена для вирусов Венесуэльского энцефалита лошадей и клещевого энцефалита на С-конце ламининсвязывающего белка человека. Биохимия, 2009, 74 (12) 1631-1641 Малыгин А.А., Бондаренко Е.И., Иванисенко В.А., Протопопова Е.В., Карпова Г.Г., Локтев В.Б. BIOCHEMISTRY-MOSCOW+, 2009, 74 (12) 1631-1641 |
|
| Выявление новых DRE в регуляторной области генов человека, кодирующих компоненты цитозольного комплекса арил-гидрокарбонового рецептора Д.Ю. Ощепков, Д.П. Фурман, Е.А. Ощепкова, А.В. Катохин, М.Ю. Шаманина, В.А. Мордвинов Информационный вестник ВОГИС, 2009, Т.13. № 1. С. 46-52. |
|
| 2008 | Functional divergence of H-pylori related to early gastric cancer Momynaliev KT, Kashin SV, Chelysheva VV, Selezneva OV. Demina IA, Serebryakova MV, Ivanisenko VA, Aman E, Akopian T, Govorun VM. HELICOBACTER, 2008, 5, 13, 477-477 |
| Regulatory region of human genes encoding macrophageal transcription factors possess multiple potential dioxin response elements Oshchepkova EA, Furman DP Oshchepkov DY, Katokhin AV, Shamanina MY, Mordvinov VA, Tsyrlov IB Organohalogen Compounds, 2008, V. 70. P. 001467-001470. |
|
| Web-based computational tools for the prediction and analysis of post-translational modifications of proteins. Ivanisenko V.A., Afonnikov D.A., Kolchanov N.A. Methods in Molecular Biology, 2008, V. 446, P. 363-384. |
|
| TRRD: Technology for extraction, storage, and use of knowledge about the structural-functional organization of the transcriptional regulatory regions in the eukaryotic genes N.A. Kolchanov, E.V. Ignatieva, O.A. Podkolodnaya, E.A. Ananko, T.M. Khlebodarova, I.L. Stepanenko, T.I. Merkulova, V.M. Merkulov, N.L. Podkolodnyy, A.G. Romashchenko INTELL DATA ANAL, 2008, No. 5, Vol. 12, P. 443-461 |
|
| uORFs, reinitiation and alternative translation start sites in human mRNAs Kochetov A.V., Ahmad S., Ivanisenko V., Volkova O.A., Kolchanov N.A., Sarai A. FEBS LETT, 2008, 582. 1293-1297 |
|
| Associative Network Discovery (AND) компьютерная система для автоматической реконструкции сетей ассоциативных знаний о молекулярно-генетических взаимодействиях П.С. Деменков, Е.Э. Аман, В.А. Иванисенко Вычислительные технологии, 2008, 2, 13, 15-19 |
|
| 2007 | Компьютерно-экспериментальный подход к дизайну полифункционального геносенсора, полученного на основе промотора гена yfiA Escherichia coli Тикунова Н.В., Хлебодарова Т.М., Крачко А.В., Степаненко И.Л., Колчанов Н.А. Doklady Akademii Nauk, 2007, 417, №6 , 835-839 |
| Detection of new potentially active DRE sites in regulatory region of human genes encoding components of Ah receptor cytosolic complex Nedosekina EA, Oshchepkov DY, Katokhin AV, Kuznetsova TN, Shamanina MY, Mordvinov VA, Tsyrlov I Organohalogen Compounds, 2007, V. 69. P. 1889-1892. |
|
| A mathematical model for the adenylosuccinate synthetase reaction involved in purine biosynthesis Evgeniya A Oshchepkova-Nedosekina, Vitalii A Likhoshvai THEOR BIOL MED MODEL, 2007, 4:11 |
|
| Mathematical model for suppression of subgenomic hepatitis C virus RNA replication in cell culture Mishchenko E.L., Bezmaternykh K.D., Likhoshvai V.A., Ratushny A.V., Khlebodarova T.M., Sournina N. Yu., Ivanisenko V.A., Kolchanov N.A. Journal of Bioinformatics and Computational Biology, 2007, Vol. 5, No. 02B pp.593-609 |
|
| Phylogenetic analysis of the p53 and p63/p73 gene families Pintus S.S., Fomin E.S., Oshurkov I.S., Ivanisenko V.A. In Silico Biology, 2007, 7(3), pp.319-332 |
|
| Web-based Computational Tools for the Prediction and Analysis of Post-translational Modifications of Proteins Ivanisenko V.A., Afonnikov D.A., Kolchanov N.A. Methods in Molecular Biology, 2007, 446, 363-384. |
|
| Application of bioinformatics resources for genosensor design Khlebodarova T.M., Tikunova N.V., Kachko A.V., Stepanenko I.L., Podkolodny N.L., Kolchanov N.A. Journal of Bioinformatics and Computational Biology, 2007, V.5. P.507-520 |
|
| Иммунохимические свойства белка PrМ и С-концевого фрагмента белка М вируса Западного Нила Богачек М.В., Протопопова Е.В., Терновой В.А., Качко А.В., Иванова А.В., Иванисенко В. А., Швалов А.Н., Локтев В.Б. Molecular Biology, 2007, 2007, т. 41(1) с. 8-17 |
|
| Иммунохимические свойства белка PrM и С концевого фрагмента белка М вируса Западного Нила Богачек М.В., Протопопова Е.В., Терновой В.А., Качко А.В., Иванова А.В., Иванисенко В.А., Швалов А.Н., Локтев В.Б. Molecular Biology, 2007, 1, 41, 8-17 |
|
| 2006 | Математическая модель репликации РНК репликона вируса гепатита С в клеточной культуре. Моделирование действия потенциальных лекарственных препаратов Безматерных К.Д., Мищенко Е.Л., Лихошвай В.А., Хлебодарова Т.М.,Иванисенко В.А. BIOPHYSICS, 2006, т. 51, выпуск N7 |
| Подтверждение функциональности дополнительного Zn2+ сайта связывания в мутантной форме G245C белка p53 Э. С. Фомин, В. А. Иванисенко BIOPHYSICS, 2006, N7, т.51 |
|
| Предсказание изменения термодинамической стабильности белков при одиночных аминокислотных заменах Деменков П.С., Аман Е.Э., Иванисенко В.А. BIOPHYSICS, 2006, Т. 7, Вып. 51 |
|
| Филогенетический анализ семейства p53 Пинтус С.С., Фомин Э.С., Иванисенко В.А., Колчанов Н.А. BIOPHYSICS, 2006, т.51, вып.4, с.640-649 |
|
| Prediction of the Changes in Thermodynamic Stability of Proteins Caused by Single Amino Acid Substitutions Demenkov P.S., Aman E.E., Ivanisenko V.A. BIOPHYSICS, 2006, т. 51, выпуск N7 |
|
| Библиотека программных компонент MOLKERN для построения программ молекулярного моделирования Фомин Э.С., Алемасов Н.А., Чирцов А.С., Фомин А.Э. BIOPHYSICS, 2006, т.51, вып.7 |
|
| 2005 | Иммунохимические свойства рекомбинантных полипептидов, моделирующих домены I и II белка Е вируса Западного Нила Богачек М.В., Протопопова Е.В., Терновой В.А., Качко А.В., Иванова А.В., Иванисенко В.А., Локтев В.Б. Molecular Biology, 2005, N 5, т. 39, с. 813-822. |
| Актуальные проблемы компьютерной протеомики Иванисенко В.А., Афонников Д.А., Николаев С.В., Пинтус С.С., Крестьянова М.А., Пальянов А.Ю, Титов И.И. Информационный вестник ВОГИС, 2005, т.9, с.162-178 |
|
| WebProAnalyst: an interactive tool for analysis of quantitative structure-activity relationships in protein families Ivanisenko V.A., Eroshkin A.M., Kolchanov N.A. NUCLEIC ACIDS RES, 2005, V. 33, W99-W104 |
|
| PDBSite: a database of the 3D structure of protein functional sites Ivanisenko VA, Pintus SS, Grigorovich DA, Kolchanov NA. NUCLEIC ACIDS RES, 2005, V33, D183–D187 |
|
| Новосибирская школа системной компьютерной биологии: исторический экскурс и перспективы развития Ратушный А.В., Лихошвай В.А., Ананько Е.А., Владимиров Н.В., Гунбин К.В., Лашин С.А., Недосекина Е.А., Николаев С.В., Омельянчук Л.В., Матушкин Ю.Г., Колчанов Н.А. Информационный вестник ВОГИС, 2005, Т.9, N2, май 2005, 232-261 |
|
| GeneNet in 2005. E.A. Ananko, N.L. Podkolodny, I.L. Stepanenko, O.A. Podkolodnaya, D.A. Rasskazov, D.S. Miginsky, V.A. Likhoshvai, A.V. Ratushny, N.N. Podkolodnaya, and N.A. Kolchanov. NUCLEIC ACIDS RES, 2005, V. 33. P. D425-D427 |
|
| 2004 | Обнаружение эмпирической аксиоматической теории в условиях шумов. Деменков П.С. Вычислительные системы, 2004 |
| Регуляция генных сетей стрессового ответа активными формами кислорода Степаненко И.Л. Экологическая генетика, 2004, Т. 2 (1). C. 4-12 |
|
| 2003 | Data Mining Techniques in Bioinformatics Zagoruiko N.G., Kolchanov N.A., Pichueva A.G., Kutnenko O.A., Borisova I.A., Kochetov A.V., Ivanisenko V.A., Nikolaev S.V., Likhoshvai V.A., Ratushnyi A.V. Pattern Recognition and Image Analysis, 2003, N. 4, V. 13, P. 550-555 |
| Application of filter technology in photomorphogenesis gene network Smirnova O.G., Stepanenko I.L. In Silico Biology, 2003, N.1-2. P. 117-125 |
|
| Apoptosis Gene Network: description in the GeneNet and TRRD databases Stepanenko I., Kolchanov N. Ann.N.Y.Acad.Sci., 2003, V.1010. P.16-18 |
|
| 2002 | Быстрый генетический алгоритм для анализа вторичной структуры РНК Титов И.И., Воробьев Д.Г., Иванисенко В.А., Колчанов Н.А. RUSS CHEM B+, 2002, N7, c. 1047-1056 |
| Локализация антигенной детерминанты белка Е вируса клещевого энцефалита, узнаваемой антигемагглютинирующими моноклональными антителами, с помощью пептидной фаговой библиотеки Локтев А.В., Кувшинов В.Н., Меламед Н.В., Иванисенко В.А., Мишин В.П., Ильичев А.А. Вопросы вирусологии, 2002, N.2, Т.47, С.31-34 |
|
| Локализация конформационного эпитопа гликопротеина gp120 ВИЧ-1, узнаваемого вируснейтрализующими моноклональными антителами 2G12 Туманова О.Ю., Кувшинов В.Н., Ильичев А.А., Некрасов Б.Г., Иванисенко В.А., Козлов А.П., Сандахчиев Л.С. Molecular Biology, 2002, N.4, Т.36, С.657-663 |
|
| GeneNet database: Description and Modeling of Gene Networks. Kolchanov N.A., Nedosekina E.A., Ananko E.A., Likhoshvai V.A., Podkolodny N.L., Ratushny A.V., Stepanenko I.L., Podkolodnaya O.A., Ignatieva E.V., Matushkin Yu.G. In Silico Biology, 2002, V.2, p. 97-110 |
|
| GeneNet: a database on structure and functional organisation of gene networks. Ananko E.A., Podkolodny N.L., Stepanenko I.L., Ignatieva E.V., Podkolodnaya O.A., Kolchanov N.A. NUCLEIC ACIDS RES, 2002, V. 30, No. 1 P. 398-401. |
|
| Transcription Regulatory Regions Database (TRRD): its status in 2002 Kolchanov NA, Ignatieva EV, Ananko EA, Podkolodnaya OA, Stepanenko IL, Merkulova TI, Pozdnyakov MA, Podkolodny NL, Naumochkin AN, Romashchenko AG NUCLEIC ACIDS RES, 2002, Jan 1;30(1):312-317 |
|
| 2001 | Интерференция генных сетей апоптоза и ответа на тепловой шок. Степаненко И.Л. Molecular Biology, 2001, Т.35(6). С. 1063-1071 |
| 2000 | Анализ чужеродных эпитопов, встроенных в НВсАg. Возможные пути решения проблемы самоорганизации химерных коровых частиц Карпенко Л.И., Иванисенко В.А., Пика И.С., Чикаев Н.А., Ерошкин А.М., Меламед Н.В., Веремейко Т.А., Ильичев А.А. Molecular Biology, 2000, N.2, Т.34, С.223-229 |
| 1999 | GENEEXPRESS: интегратор баз данных и компьютерных систем, доступных по сети Интернет и предназначенных для изучения экспресии генов эукариот Н.А.Колчанов, М.П.Пономаренко, А.Э.Кель, Ю.В.Кондрахин, А.С.Фролов, Ф.А.Колпаков, Т.Н.Горячковская, О.В.Кель, Е.А.Ананько, Е.В.Игнатьева, О.А.Подколодная, И.Л.Степаненко, Т.И.Меркулова, В.В.Бабенко, Д.В.Воробьев, С.В.Лаврюшев, Ю.В.Пономаренко, А.В.Кочетов, Г.Н.Колесов, Н.Л.Подколодный, Л.Миланези, Э.Вингендер, Т.Хейнемайер, В.В.Соловьев, Г.К.Овертон BIOPHYSICS, 1999, 5, 44, 837-841 |
| Integrated databases and computer systems for studying eukaryotic gene expression. Kolchanov N.A., Ponomarenko M.P., Frolov A.S., Ananko E.A., Kolpakov F.A., Ignatieva E.V., Podkolodnaya O.A., Goryachkovskaya T.N., Stepanenko I.L., Merkulova T.I., Babenko V.N., Ponomarenko J.V., Kochetov A.V., Podkolodny N.L., Vorobyev D.G., Lavrushev S.V., Grigorovich D.A., Kondrakhin Yu.V., Milanesi L., Wingender E., Solovyev V.V., Overton G.C. BIOINFORMATICS, 1999, 7/8, 15, 669-686 |
|
| 1998 | GeneExpress: a computer system for description, analysis, and recognition of regulatory sequences in eukaryotic genome. N.A. Kolchanov, M.P. Ponomarenko, A.E. Kel, A.S. Frolov, F.A. Kolpakov, T.N. Goryachkovsky, O.V. Kel, E.A. Ananko, E.V. Ignatieva, O.A. Podkolodnaya, V.N. Babenko, I.L. Stepanenko, A.G. Romashchenko, T.I. Merkulova, D.G. Vorobiev, S.V. Lavryushev, A.V. Kochetov, G.B. Kolesov, V.V. Solovyev, L. Milanesi, N.L. Podkolodny, E. Wingender, T. Heinemeyer Proc Int Conf Intell Syst Mol Biol, 1998, 6, 95-104 |
| 1997 | Поиск сайтов, содержащих функционально важные замены в наборах родственных или мутантных белков Иванисенко В.А., Ерошкин А.М. Molecular Biology, 1997, N.5, Т.31, С.880-887. |
| Использование фаговой библиотеки пептидов в картировании группоспецифического гемагглютинирующего домена гликопротеина Е2 альфа-вирусов Кузьмичева Г.А., Кувшинов В.Н., Разумов И.А., Иванисенко В.А., Ерошкин А.М., Мишин В.П., Ушакова Т.А., Локтев В.Б., Ильичев А.А. MOL GENET MICROBIOL+, 1997, N.4, С.25-29 |
|
| Экспрессия фрагментов гена Е вируса японского энцефалита в клетках Escherichia coli Белавин П.А., Нетесова Н.А., Решетников С.С., Иванисенко В.А., Ерошкин А.М., Протопопова Е.В., Локтев В.Б., Малыгин Э.Г. Биотехнология, 1997, N.3, С.3-10 |
|
| 1996 | Компьютерный анализ зависимостей между максимумом цвета биолюминесценции и первичной структурой люцифераз жуков: идентификация сайтов, ответственных за изменения цвета Морозов В.М., Иванисенко В.А., Ерошкин А.М., Угарова Н.Н. Molecular Biology, 1996, Т.30, С.1167-1172 |
| 1994 | Получение мутантного фактора некроза опухолей человека с повышенной устойчивостью к протеазам Корепанова А.В., Ерошкин А.М., Иванисенко В.А., Лебедев Л.Р., Микрюков Н.Н., Пустошилова Н.М., Петренко В.А., Ильичев А.А. Molecular Biology, 1994, Т.28., С.143-149 |
| 1993 | Анализ встроек пептидных фрагментов в основной белок оболочки бактериофагов М13, f1 и fd. Взаимосвязь структурных характеристик белка оболочки и жизнеспособности мутантных фагов Ерошкин А.М., Миненкова O.O., Фомин В.А., Иванисенко В.А., Ильичев A.A. Molecular Biology, 1993, Т.27. С.1345-1355 |
 Монографии
Монографии
| 2021 | The Web Platform for Storing Biotechnologically Significant Properties of Bacterial Strains Mukhin A. M., Kazantsev F.V., Klimenko A.I., Lakhova T.N., Demenkov P.S., Lashin S.A. |
| 2019 | Web-Based Computational Tools for the Prediction and Analysis of Posttranslational Modifications of Proteins Ivanisenko, V. A., Ivanisenko, T. V., Saik, O. V., Demenkov, P. S., Afonnikov, D. A. and Kolchanov, N. A. |
| 2017 | Heat Shock Proteins Ponomarenko M., Stepanenko I., Kolchanov N. |
| 2016 | Создание малых химических соединений, направленно действующих на внешний путь апоптоза с помощью компьютерного моделирования и экспериментальных подходов. И.Н. Лаврик, Л. Хиллерт, Н.В. Иванисенко, О.В. Попик, О.В. Сайк, Т.В. Иванисенко, П.С. Деменков, Е.Д. Петровский, В.А. Иванисенко |
| 2015 | Молекулярное моделирование и создание малых химических соединений, направленных на регуляцию программируемой клеточной гибели, индуцируемой через клеточные рецепторы / отв. ред. И.Н. Лаврик, В.А. Иванисенко ИН Лаврик, Л Хиллерт, С Питкевич, НВ Иванисенко, ОВ Попик, ТВ Иванисенко, ПС Деменков, ЕД Петровский, ВА Иванисенко |
| 2014 | Text Mining on PubMed Timofey V. Ivanisenko, Pavel S. Demenkov, Vladimir A. Ivanisenko |
| 2013 | Heat Shock Proteins. Ponomarenko M, Stepanenko I., Kolchanov N |
| 2012 | Разработка методов, алгоритмов и программ параллельного моделирования в биоинформатике Н.Л. Подколодный, Д.А. Афонников, К.В. Гунбин, М.А. Генаев, Ю.Л. Орлов, Е.В. Игнатьева, О.В. Вишневский, В.А. Иванисенко, П.С. Деменков, Н.А. Колчанов |
| 2010 | German/Russian Network of Computational Systems Biology. R. Hofestadt and N. Kolchanov (Eds). E.L. Mishchenko, N.V. Ivanisenko, A.M.Mishchenko, V.A.Likhoshvai, V.A. Ivanisenko, R. Hofestadt, N.A. Kolchanov |
| Nandbook of Drug Targeting and Monitoring. B.Andreev and V. Egorov ( Eds). E.L. Mishchenko, K.D. Bezmaternykh, V.A. Likhoshvai, V.A. Ivanisenko, N.A. Kolchanov |
|
| Visualization and Analysis of Biological Networks combining Text Mining and Data Warehouse Approaches Björn Sommer, Evgeny S. Tiys, Benjamin Kormeier, Klaus Hippe, Sebastian J. Janowski, Timofey V. Ivanisenko, Anatoly O. Bragin, Patrizio Arrigo, Pavel S. Demenkov, Alexey V. Kochetov, Vladimir A. Ivanisenko, Nikolay A. Kolchanov and Ralf Hofestädt |
|
| 2009 | Viral Genomes: Diversity, Properties and Parameters. Zhi Feng and Ming Long (Eds). E.L. Mishchenko, K.D. Bezmaternykh, V.A. Likhoshvai, V.A. Ivanisenko, N.A. Kolchanov |
| Implementation of a Non-bonded Interaction Calculation Algorithm for the Cell Architecture Fomin E.S., Alemasov N.A. |
|
| 2008 | Регуляторные последовательности ДНК: описание в базах данных. Колчанов Н.А., Подколодная О.А., Ананько Е.А., Игнатьева Е.В., Степаненко И.Л., Хлебодарова Т.М., Меркулова Т.И., Меркулов В.М., Мищенко Е.Л., Ибрагимова С.С., Смирнова О.Г., Подколодный Н.Л., Ромащенко А.Г., Ощепков Д.Ю., Мигинский Д.С. |
| Cистемная компьютерная биология. Ред. Колчанов Н.А., Гончаров С.С., Лихошвай В.А., Иванисенко В.А., Изд-во СО РАН, Новосибирск Хлебодарова Т.М., Тикунова Н.В., Качко А.В., Степаненко И.Л., Колчанов Н.А. |
|
| Методы моделирования динамики молекулярно-генетических систем Лихошвай В.А., Ратушный А.В., Бажан. С.И., Ощепкова Е.А., Фадеев С.И., Хлебодарова Т.М., Колчанов Н.А. |
|
| «Системная компьютерная биология». Отв. Редакторы: Н.А.Колчанов, С.С.Гончаров., В.А. Лихошвай, В.А. Иванисенко , Н: Изд. СО РАН, , 2008, 761с Н.А.Колчанов, С.С.Гончаров., В.А. Лихошвай, В.А. Иванисенко |
|
| Распознавание функциональных сайтов в пространственных структурах белков Иванисенко В.А., Деменков П.С., Фомин Э.С., Крестьянова М.А., Ощурков И.С., Иванисенко Т.В., Иванисенко Н.В., Пинтус С.С., Яркова Е.Э., Степаненко И.Л., Сурнина Н.Ю., Гончаров С.С., Колчанов Н.А. |
|
| IV Международная конференция "Фундаментальные науки - медицине". Сборник трудов конференции. Е.Л. Мищенко, К.Д.Безматерных, В.А. Лихошвай, В.А. Иванисенко |
|
| Филогенетический и структурный анализ семейства p53 Пинтус С.С., Фомин Э.С., Иванисенко В.А. |
|
| 2006 | TRANSCRIPTION REGULATORY REGIONS DATABASE (TRRD): A SOURCE OF EXPERIMENTALLY CONFIRMED DATA ON TRANSCRIPTION REGULATORY REGIONS OF EUKARYOTIC GENES N. Kolchanov, E. Ignatieva, O. Podkolodnaya, E. Ananko, I. Stepanenko, T. Merkulova, T. Khlebodarova, V. Merkulov, N. Podkolodny, D. Grigorovich, A. Poplavsky, A. Romashchenko |
| STUDY OF THE INTERACTIONS BETWEEN VIRAL AND HUMAN GENOMES DURING TRANSFORMATION OF B CELLS WITH EPSTEIN-BARR VIRUS E. Ananko, D. Oshchepkov, E. Nedosekina, V. Levitsky, I. Lokhova, O. Smirnova, V. Likhoshvai, N. Kolchanov. |
 Конференции
Конференции
| 2025 | Integration of bioinformatics data for crop plant breeding. Yuriy L. Orlov, Haoyu Chao, Shilong Zhang, Vladimir A. Ivanisenko, Ming Chen. BelBI-2024 |
| 2024 | Transfer learning based yeast protein abundance prediction Venzel A.S., Klimenko A.I., Demenkov P.S., Ivanisenko T.V., Lashin S.A., Kolchanov N.A., Ivanisenko V.A. 14th International Multiconference Bioinformatics of Genome Regulation and Structure/Systems Biology |
| 2022 | Генетические исследования и клиническая лимфология Нимаев В.В. Сайк О.В. XIV Научно-практическая конференция Ассоциации флебологов России с международным участием «Актуальные вопросы флебологии» |
| On organizing a software platform for seeking
biotechnologically important features in bacteria Lashin S., Demenkov P., Ivanisenko V., Kazantsev F., Mukhin A., Kolchanov N BGRS/SB-2022: 13th International Multiconference on “Bioinformatics of Genome Regulation and Structure/Systems Biology” |
|
| Data lake platform of the bacterial strain properties
for microbiology Mukhin A., Demenkov P., Kazantsev F., Lakhova T., Klimenko A., Lashin S BGRS/SB-2022: 13th International Multiconference on “Bioinformatics of Genome Regulation and Structure/Systems Biology” |
|
| 2021 | A feedback loop enrchment analysis in gene network of bronchial asthma and pulmonary tuberculosis interaction Evgeny S. Tiys, Pavel S. Demenkov, Vladimir A. Ivanisenko, Nikolay A. Kolchanov BGRS/SB-2020: 12th International Multiconference “Bioinformatics of Genome Regulation and Structure/Systems Biology” |
| ВЫЯВЛЕНИЕ ГЕНОВ, АССОЦИИРОВАННЫХ ОДНОВРЕМЕННО С ОЖИРЕНИЕМ И ЛИМФЕДЕМОЙ, С ПОМОЩЬЮ СИСТЕМЫ ANDSYSTEM Сайк О.В., Деменков П.С., Иванисенко В.А., Нимаев В.В. Международная научно-практическая конференция «Лимфология: от фундаментальных исследований к медицинским технологиям» |
|
| Гены сигнального пути IL-6, ассоциированные с диабетической ретинопатиейи вариабельностью гликемии Сайк О.В., Климонтов В.В. IV Российская междисциплинарная научно-практическая конференция с международным участием «Сахарный диабет: от мониторинга к управлению» |
|
| Text Mining and Bioinformatic Identification of Genes Linking Glycemic Variability and Impaired Angiogenesis in Diabetes Olga V. Saik, Vadim V. Klimontov 81st Scientific Sessions of American Diabetes Association (ADA) |
|
| Bioinformatic analysis by ANDSystem revealed members of IL-6 signaling pathway initiated by glucose variability and involved in diabetic retinopathy Olga Saik, Vadim Klimontov 2021 IEEE Ural-Siberian Conference on Computational Technologies in Cognitive Science, Genomics and Biomedicine (CSGB) |
|
| The Web Platform for Storing Biotechnologically Significant Properties of Bacterial Strains Aleksey M. Mukhin , Fedor V. Kazantsev, Alexandra I. Klimenko, Tatiana N. Lakhova, Pavel S. Demenkov, and Sergey A. Lashin International Conference on Parallel Computing Technologies |
|
| 2020 | ANDDIGEST: A TEXT-MINING BASED COMPUTER SYSTEM FOR GENERATING DIGESTS IN THE FIELD OF BIOLOGY IVANISENKO TIMOFEY, DEMENKOV PAVEL, IVANISENKO VLADIMIR, KOLCHANOV NIKOLAY BGRS/SB-2020. THE 12TH INTERNATIONAL MULTICONFERENCE |
| Learning the changes of barnase mutants thermostability from
structural fluctuations obtained using anisotropic network
modeling Nikolay Alemasov, Nikita Ivanisenko, Vladimir Ivanisenko Twelfth International Multiconference BGRS/SB-2020 |
|
| Interpretation of the features of a linear regression model
for predicting the survival time of the amyotrophic lateral
sclerosis patients with mutated SOD1 Nikolay Alemasov, Alexandr Shcherbakov, Vladimir Timofeev, Vladimir Ivanisenko Twelfth International Multiconference BGRS/SB-2020 |
|
| Выявление злокачественных опухолей щитовидной железы в мазках с неопределенным цитологическим заключением с помощью молекулярного классификатора. Титов СЕ, Иванов МК, Веряскина ЮА, Деменков ПС, Шевченко СП, Катанян ГА, Лукьянов СА. VI ПЕТЕРБУРГСКИЙ МЕЖДУНАРОДНЫЙ ОНКОЛОГИЧЕСКИЙ ФОРУМ "БЕЛЫЕ НОЧИ 2020» |
|
| Melatonin as a key regulator in molecular-genetic network of glucose variability related to circadian rhythm Cайк О., Деменков П., Иванисенко В., Климонтов В. BGRS/SB-2020: 12th International Multiconference “Bioinformatics of Genome Regulation and Structure/Systems Biology”,Symposium “Systems biology and biomedicine” (SbioMed-2020) |
|
| EXTRACTION OF SPECTRAL SERIES OF IONS FROM MASS SPECTRA OF PEPTIDES BY METHODS OF INTEGRAL TRANSFORMS AND MACHINE LEARNING Fomin E., Alemasov N., Afonnikov D. BGRS-2020 |
|
| Выделение спектральных серий в тандемных масс спектрах с помощью машинного обучения Фомин Э.С., Алемасов Н. Международная конференция «Марчуковские научные чтения 2020», МНЧ-2020 |
|
| Выявление ключевых участников молекулярно-генетических сетей, ассоциированных с лимфедемой, с помощью биоинформатических методов Сайк О.В., Деменков П.С., Иванисенко В.А., Нимаев В.В. 7-й съезд лимфологов России и 8-я международная научно-практическая конференция по клинической лимфологии "ЛИМФА-2020" |
|
| Компьютерное моделирование влияния циркадных часов на воспалительную реакцию
на бактериальную инфекцию Н.Л. Подколодный, Н.Н. Твердохлеб, О.А. Подколодная Международная конференция «Марчуковские научные чтения 2020» (МНЧ-2020) |
|
| Functional annotation of the transcription factors from Methylotuvimicrobium alcaliphilum 20ZR. Kolmykov S.K., Kulyashov M., Ivanisenko N.V., Evshin I.S., Khlebodarova T.M., Akberdin I.R. 12th International Multiconference “Bioinformatics of Genome Regulation and Structure/Systems Biology” (BGRS/SB-2020) |
|
| Integration of transcriptomics data into a genome-scale metabolic model of the methanotrophic bacterium Methylotuvimicrobium alcaliphilum 20ZR. Kolmykov S.K., Kulyashov M., Ivanisenko N.V., Evshin I.S., Khlebodarova T.M., Akberdin I.R. 12th International Multiconference “Bioinformatics of Genome Regulation and Structure/Systems Biology” (BGRS/SB-2020) |
|
| 2019 | НАСЛЕДУЕМЫЕ БАКТЕРИАЛЬНЫЕ СИМБИОНТЫ НАСЕКОМЫХ: РАЗНООБРАЗИЕ И ЭВОЛЮЦИЯ Илинский Ю.Ю., Быков Р.А., Деменкова М.А., Юрлова Г.В., Деменков П.С., Брошков А.Д., Данилова М.В., Вяткин Ю.В. VII съезд Вавиловского общества генетиков и селекционеров, посвященный 100-летию кафедры генетики СПбГУ, и ассоциированные симпозиумы. 18-22 июня 2019г, Санкт-Петербург. |
| RECONSTRUCTION OF ASSOCIATIVE GENE NETWORKS BASED ON AUTOMATIC EXTRACTION OF KNOWLEDGE FROM SCIENTIFIC PUBLICATIONS, PATENTS AND DATABASES USING INTELLIGENCE METHODS VA Ivanisenko, ES Tiys, TV Ivanisenko, PS Demenkov VII съезд Вавиловского общества генетиков и селекционеров, посвященный 100-летию кафедры генетики СПбГУ, и ассоциированные симпозиумы. 18-22 июня 2019г, Санкт-Петербург. |
|
| РЕКОНСТРУКЦИЯ И АНАЛИЗ ГЕННОЙ СЕТИ ВЗАИМОДЕЙСТВИЙ БОЛЕЗНИ АЛЬЦГЕЙМЕРА И ВИРУСНЫХ ПРОЦЕССОВ: ПОИСК НОВЫХ ГЕНОВ, ВОВЛЕЧЁННЫХ В МЕХАНИЗМ ЗАБОЛЕВАНИЯ Тийс Е.С., Иванисенко В.А. VII съезд Вавиловского общества генетиков и селекционеров, посвященный 100-летию кафедры генетики СПбГУ, и ассоциированные симпозиумы |
|
| Dysregulation of postsynaptic translation in autism spectrum disorder: reconstruction of gene network associated with autism and mtor signaling. Rifonova E.A., Klimenko A.I., Saik O.V., Orlov Y.L., Lashin S.A., Ivanisenko V.A. VII съезд Вавиловского общества генетиков и селекционеров, посвященный 100-летию кафедры генетики СПбГУ, и ассоциированные симпозиумы. 18-22 июня 2019г, Санкт-Петербург. |
|
| 2018 | Поиск генов, потенциально ассоциированных с лимфедемой, из числа генов, вовлеченных в эндотелиальный апоптоз, с использованием анализа ассоциативных генных сетей ANDSYSTEM Сайк О.В., Нимаев В.В., Усмонов Д.А., Деменков П.С., Иванисенко Т.В., Лаврик И.Н., Иванисенко В.А. XIII международная научно-практическая конференция «Лимфология: от фундаментальных исследований к медицинским технологиям» |
| Analysis of Clinical Forms of Leg Lymphedema. Single-Center Experience D. Usmonov, O. Saik, V. Nimaev Bioinformatics of Genome Regulation and Structure\ Systems Biology — BGRS\SB-2018 Symposium Systems Biology and Biomedicine (SBioMed-2018) |
|
| Topological properties of graph of hydrogen bonds forming in SOD1 protein indicate critical regions in its structure N. Alemasov, V. Ivanisenko Systems Biology of DNA Repair Processes and Programmed Cell Death (SbPCD-2018) |
|
| Rational design of molecular probes targeting caspase-8/c-FLIPL heterodimer in the Death-Inducing Signaling Complex Иванисенко Никита Владимирович 26th Conference of the European Cell Death Organization |
|
| Study of bioresources of salt lakes of Novosibirsk oblast A. Bryanskaya, Yu. Uvarova, A. Rozanov, E. Lazareva, O. Taran, T. Ivanisenko, S. Peltek 11th International Multiconference “Bioinformatics of Genome Regulation and Structure\Systems Biology” — BGRS\SB-2018 |
|
| Reconstruction of Gene Networks Associated with Autism and Related to mTOR Signaling Pathway using ANDSystem O.V. Saik, E.A. Trifonova, T.M. Khlebodarova, V.A. Ivanisenko BGRS\SB-2018 |
|
| Analysis of Programmed Cell Death in Associative Gene Network of Glaucoma Reconstructed Using ANDSystem O.V. Saik, P.S. Demenkov, O.S. Konovalova, N.A. Konovalova, I.N. Lavrik, V.A. Ivanisenko BGRS\SB-2018 |
|
| ПРОВЕРКА ВЫПОЛНЕНИЯ СВОЙСТВА ТРАНЗИТИВНОСТИ В ФРЕЙМОВЫХ МОДЕЛЯХ, ОПИСЫВАЮЩИХ СВЯЗЬ ГЕНЕТИЧЕСКОЙ РЕГУЛЯЦИИ МЕТАБОЛИЧЕСКИХ ПРОЦЕССОВ С ЗАБОЛЕВАНИЯМИ Сайк О.В., Деменков П.С., Иванисенко В.А. Международная научно-практическая конференция «Экспериментальные и теоретические исследования в современной науке» |
|
| Comparison of quality of automated gene network reconstruction
using connectivity of random and functional networks Тийс Е.С., Деменков П.С., Иванисенко В.А. Mathematical Modeling and High-Performance Computing in Bioinformatics, Biomedicine and Biotechnology (MM-HPC-BBB-2018) |
|
| 2017 | A METAGENOMIC STUDY OF BENTHIC MICROBIAL COMMUNITIES OF SALINE LAKES OF THE NOVOSIBIRSK OBLAST BRYANSKAYA A.V., UVAROVA YU. E., ROZANOV A.S., MALUP T.K., LAZAREVA E.V., TARAN O.P., DAGUROVA O.P., IVANISENKO Т.V., PELTEK S.E. 13TH INTERNATIONAL CONFERENCE ON SALT LAKE RESEARCH (ICSLR 2017) Ulan-Ude, Russia, 21-25 августа 2017 г. |
| Раскрытие общих механизмов агрегации мутантов белка SOD1 с помощью эластичного сетевого моделирования Н.А. Алемасов, В.А. Иванисенко Сателлитный симпозиум «Молекулярно-генетические механизмы патогенеза глаукомы» |
|
| ANDSystem: a tool for automated literature mining in the field of biology Ivanisenko V.A., Saik O.V., Tiys E.S., Ivanisenko T.V., Demenkov P.S. Международный конгресс "Биотехнология: состояние и перспективы развития" |
|
| Сахарный диабет в эпоху «омиксных технологий» О.В. Сайк, П.С. Деменков, Н.А. Колчанов, В.А. Иванисенко II Междисциплинарная конференция "Сахарный диабет-2017: от мониторинга к управлению" |
|
| RECONSTRUCTION OF ASSOCIATIVE GENE NETWORK RELATED TO AGGRESSIVE BEHAVIOR IN RATS USING TRANSCRIPTOME DATA O.V. Petrovskaya, I.V.Chadaeva, O.V. Saik, A.O. Bragin, E.S. Tyis БЕЛЯЕВСКИЕ ЧТЕНИЯ, Международная конференция, посвященная 100-летию со дня рождения академика АН СССР Д.К. Беляева |
|
| Реконструкция генных сетей, ассоциированных с первичной открытоугольной глаукомой с помощью системы ANDSystem. Сайк О.В., Деменков П.С., Коновалова Н.А., Коновалова О.С., Иванисенко В.А. Беляевские чтения: Международная конференция, посвященная 100-летию со дня рождения академика АН СССР Д.К. Беляева |
|
| Analysis of microRNA expression in rat pluripotent stem cells using genome-wide sequencing technologies Sherstyuk V.V., Medvedev S.P., Elisaphenko E.A., Vaskova E.A., Ri M.T., Vyatkin Y.V., Saik O.V., Shtokalo D.N., Pokushalov E.A., Zakian S.M. 15th Annual Meeting of the International Society for Stem Cell Research |
|
| ПОИСК ГЕНОВ, ОТВЕТСТВЕННЫХ ЗА КОМОРБИДНОСТЬ ПЕРВИЧНОЙ ОТКРЫТО-УГОЛЬНОЙ ГЛАУКОМЫ И ДИАБЕТИЧЕСКОЙ РЕТИНОПАТИИ, С ПОМОЩЬЮ СТРУКТУРНОГО АНАЛИЗА ГЕННЫХ СЕТЕЙ Е.С. Тийс, О.В. Сайк, В.А. Иванисенко Беляевские чтения: междунар. конф., посвященная 100-летию со дня рождения акад. АН СССР Д.К.Беляева |
|
| 2016 | Computer Analysis of RNA-seq Data of Laboratory Rats with Aggressive Behavior Anatoly O. Bragin*, Vladimir N. Babenko, Anastasia M. Spitsina, Ekaterina V. Kulakova, Irina V. Medvedeva, Irina V. Chadaeva, Yuriy L. Orlov, Arcady L. Markel SocBin Bioinformatics 2016 |
| SEARCH FOR GENE MUTATIONS THAT CAN POTENTIALLY AFFECT THE SUSCEPTIBILITY TO TUBERCULOSIS O.V. Saik, P.S. Demenkov, E.U. Bragina, M. Freidin, A. El-Seedy, R. Hofestaedt, V.A. Ivanisenko BGRS\SB-2016 |
|
| AIMEDICA - INTELLIGENT SYSTEM FOR DISEASE DIAGNOSTICS BASED ON TEXT-MINING ANALYSIS OF SCIENTIFIC PUBLICATIONS AND DIFFERENT MEDICAL DATA SOURCES O.V. Saik, P.S. Demenkov, A.V. Starkov, T.V. Ivanisenko, E.V, Gaisler, V.A. Ivanisenko BGRS\SB-2016 |
|
| ASSOCIATIVE NETWORKS OF GLAUCOMA AND APOPTOSIS O.V. Saik, P.S. Demenkov, O.S. Konovalova, M.N. Ponomareva, N.A. Konovalova, N.A. Kolchanov, I.N. Lavrik, V.A. Ivanisenko BGRS\SB-2016 |
|
| Reconstruction of molecular-genetic networks common for Alzheimer's disease O.V. Saik, V.A. Ivanisenko, P.S. Demenkov, E.I. Rogaev BGRS\SB-2016 |
|
| Approach to predicting the solubility/insolubility of E. coli proteins based on their primary structure using sequence normalization and machine learning techniques N.A. Alemasov, N.V. Ivanisenko, K.S. Antonets, A.A. Nizhnikov, V.A. Ivanisenko BGRS\SB-2016 |
|
| Gene ontology analysis and network reconstruction for genes related to aging diseases and behavior I.V. Chadaeva, O.V. Saik, V.N. Babenko BGRS 2016 |
|
| Analysis of aggressive behavior by whole RNA sequencing in brain compartments V.N. Babenko, A.O. Bragin, I.V. Medvedeva, I.V. Chadaeva, A.I. Dergilev, A.M. Spitsyna, N.N. Kudriavtseva, A.L. Markel, Y.L. Orlov Нейроинформатика 2016 |
|
| Microbial community of the oil site of the Uzon caldera (Kamchatka) S.E. Peltek, A.V. Bryanskaya, Y.E. Uvarova, A.S. Rozanov, T.V. Ivanisenko, T.K. Malup, V.A. Ivanisenko, E.V. Lazareva, O.V. Saik, S.M. Zhmodik, O.P. Taran, N.M. Slynko, S.V. Shekhovtsov, V.N. Parmon, N.L. Dobretsov, N.A. Kolchanov 10th anniversary International Multiconference «Bioinformatics of Genome Regulation and Structure/Systems Biology» |
|
| Микробные сообщества экстремальных экосистем; метагеномный подход С.Е. Пельтек, А.В. Брянская, Ю.Е. Уварова, А.С. Розанов, Т.В. Иванисенко, Т.К. Малуп, В.А. Иванисенко, Е.В. Лазарева, О.В. Сайк, С.М. Жмодик, О.П. Таран, Н.М. Слынько, С.В. Шеховцов, В.Н. Пармон, Н.Л. Добрецов, Н.А. Колчанов V Съезд биохимиков России |
|
| Microbial Community of the Extremal Ecosystems of the Uzon Caldera (Kamchatka) S.E. Peltek, A.V. Bryanskaya, Y.E. Uvarova, A.S. Rozanov, T.V. Ivanisenko, T.K. Malup, V.A. Ivanisenko, E.V. Lazareva, O.V. Saik, V.M. Efimov, S.M. Zhmodik, O.P. Taran, N.M. Slynko, S.V. Shekhovtsov, V.N. Parmon, N.L. Dobretsov, N.A. Kolchanov 7th World Congress onMicrobiology |
|
| How are protein functional sites encoded by exon structure in Metazoas Медведева И.В., Деменков П.С., Иванисенко В.А. SocBin Bioinformatics 2016 |
|
| GeneOntology biological processes sensitive to salt diet changes in an expreiment with 105-day isolation: statistical analysis of urine proteome E.S. Tiys, E.D. Petrovskiy, L.Kh. Pastushkova, D.N. Kashirina, I.M. Larina, V.A. Ivanisenko BGRS/SB’2016 |
|
| ANDSystem: система автоматического извлечения знаний и интеграции омиксных данных. В.А. Иванисенко, О.В. Сайк, Н.В. Иванисенко, Т.В. Иванисенко, П.С. Деменков, Н.А. Колчанов V СЪЕЗД ФИЗИОЛОГОВ СНГ и V СЪЕЗД БИОХИМИКОВ РОССИИ |
|
| Novel approach for computational design of small molecule inhibitors of protein/protein interactions in CD95/FAS pathway Nikita Ivanisenko, A.S. Ishchenko, I.N. Lavrik, V.A. Ivanisenko BGRS\SB-2016 |
|
| MOSAIC GENE NETWORK MODELLING IDENTIFIED NEW REGULATORT MEChANISMS IN HCVINFECTION O.V. Petroskaya, E.D. Petrovskiy, E.L. Mishchenko, I.N. Lavrik, V.A. Ivanisenko BGRS\SB-2016 |
|
| The compendium of human genes controlling feeding behavior or body weight, reconstruction of networks and analysis of their properties. Ignatieva E.V., O.V. Saik, D.A. Afonnikov The Tenth International Conference On Bioinformatics of Genome Regulation and Structure\Systems biology BGRS/SB2016 |
|
| Thermophilic Microbial Community of the Extremal Ecosystems S.E. Peltek, A.V. Bryanskaya, Y.E. Uvarova, A.S. Rozanov, T.V. Ivanisenko, T.K. Malup, V.A. Ivanisenko, E.V. Lazareva, O.V. Saik, V.M. Efimov, S.M. Zhmodik, O.P. Taran, N.M. Slynko, S.V. Shekhovtsov, V.N. Parmon, N.L. Dobretsov, N.A. Kolchanov The Fifth International Conference on Sustainable Utilization of Tropical Plant Biomass - Bioproducts, Biocatalysts, and Biorefinery (SUTB4), 17th to 18th November 2016 Coimbatore, Tamilnadu, India |
|
| 2015 | Молекулярно-динамическая модель для оценки влияния мутаций в белке на фенотипические признаки организма Алемасов Н.А., Иваниесенко Н.В., Иванисенко В.А. Актуальные проблемы вычислительной и прикладной математики 2015 (AMCA 2015) |
| Применение методов анализа текстов для построения онтологий Деменков П.С., Иванисенко Т.В., Сайк О.В., Иванисенко В.А. "Знания-Онтологии-Теории" 2015 |
|
| Автоматический анализ текстов в задачах биологии и медицины Деменков П.С., Иванисенко Т.В., Сайк О.В., Иванисенко В.А. Форум – 2015: «Big Data в медицине: лучшие практики» |
|
| ANDSystem: automated literature mining and protein interactome networks reconstruction Saik O.V., Demenkov P.S., Tiys E.S., Ivanisenko T.V., Kolchanov N.A., Ivanisenko V.A. VII РОССИЙСКИЙ СИМПОЗИУМ «БЕЛКИ И ПЕПТИДЫ» |
|
| Promedia a database containing frame models of genetic regulation of the enzymatic processes associated with diseases Saik O.V., Demenkov P.S., Ivanisenko V.A. VII РОССИЙСКИЙ СИМПОЗИУМ «БЕЛКИ И ПЕПТИДЫ» |
|
| Структурные и динамические особенности мутантов белка SOD1, ассоциированных с боковым амиотрофическим склерозом Алемасов Н.А., Иванисенко Н.В., Иванисенко В.А. Белки и пептиды 2015 |
|
| Модель сортов пшениц нового поколения: скороспелость и несимбиотическая азотфиксация Гончаров Николай Петрович, Степаненко Ирина Лембитовна, Ефимов Вадим Михайлович международная конференция “Генетическая интеграция прокариот и эукариот: фундаментальные исследования и современные агротехнологии” |
|
| “Application of ANDSystem for the reconstruction and analysis of molecular-genetics networks, associated with diseases and phenotypic traits” (“Применение ANDSystem для реконструкции и анализа молекулярно-генетических сетей, связанных с фенотипическими признаками и развитием заболеваний”) О.В. Сайк, П.С. Деменков, Т.В. Иванисенко, Е.С. Тийс, В.А. Иванисенко. СИМБИОЗ – РОССИЯ 2015 (VIII Всероссийский с международным участием Конгресс молодых ученых-биологов) |
|
| Контекстный анализ научных публикаций по биологии и извлечение знаний В. А. Иванисенко, Т.В. Иванисенко, П. С. Деменков, О.В. Сайк Актуальные проблемы вычислительной и прикладной математики 2015 (AMCA 2015) |
|
| Computer Tools for Gene Expression Data Processing and Correlation Analysis Babenko V.N., Orlov Y. L., Spitsyina A. M. Podkolodnaya N. Computational systems biology and bioinformatics (CSBio 2015) |
|
| Использование пациент-специфичных индуцированных плюрипотентных стволовых клеток для моделирования синдрома удлиненного интервала QT Дементьева Е.В., Григорьева Е.В., Вялкова А.В., Медведев С.П., Шевченко А.И., Елисафенко Е.А., Байрамова С.А., Иванисенко Н.В., Иванисенко В.А., Покушалов Е.А., Караськов А.М., Закиян С.М. VI Всероссийский съезд аритмологов |
|
| ИСПОЛЬЗОВАНИЕ КОМПЬЮТЕРНОЙ ТЕХНОЛОГИИ GeneNet ДЛЯ РЕКОНСТРУКЦИИ ФРАГМЕНТОВ ГИБРИДНОЙ ГЕННОЙ СЕТИ СИМБИОТИЧЕСКОЙ АЗОТФИКСАЦИИ Ибрагимова С.М., Степаненко И.Л. Фундаментальные и прикладные проблемы современной экспериментальной биологии растений |
|
| Системная компьютерная биология: анализ и моделирование структурно- функциональной организации и эволюции генных сетей Н.Л. Подколодный, Д.А. Афонников, В.А. Иванисенко, Ю.Г. Матушкин, Н.А. Колчанов Международная конференция «Актуальные проблемы вычислительной и прикладной математики 2015» |
|
| Gene Expression Analysis in Brain Areas of Laboratory Rats with Aggressive Behavior by RNA-seq Data Anatoly O. Bragin, Vladimir N. Babenko, Anastasia M. Spitsina, Ekaterina V. Kulakova, Irina V. Medvedeva, Irina V. Chadaeva, Yuriy L. Orlov, Arcady L. Markel CSBio’2015 |
|
| 2014 | Computer analysis of expression level of allergen-coding genes of pathogenic microorganisms Bragin A.O.,Sokolov V.S., Demenkov P.S., Matushkin Yu.G., Ivanisenko V.A. BGRS\SB-2014 |
| ANDSystem: associative network discovery system for automated literature mining in the area of biology V.A. Ivanisenko, O.V. Saik, E.S. Tiys, T.V. Ivanisenko, P.S. Demenkov Bioinformatics of Genome Regulation and Structure\Systems Biology — BGRS\SB-2014 |
|
| Mathematical modeling of the interactions between molecular genetic systems based on the data about perturbations of the systems elements Popik O.V., Kolchanov N.A., Ivanisenko V.A 9th International Conference on Bioinformatics of Genome Regulation and Structure |
|
| Evaluation of pathways’ efficiency based on data on PPI and distribution of proteins over cellular localizations. Popik O.V., Hofestaedt R., Ivanisenko V.A. 9th International Conference on Bioinformatics of Genome Regulation and Structure |
|
| L-MOLKERN software allowing for polarization effects in free energy calculation E.S. Fomin, N.A. Alemasov BGRS/SB-2014 |
|
| Structural and dynamical properties of SOD1 protein mutants related to familial amyotrophic lateral sclerosis Nikolay Alemasov, Nikita Ivanisenko, Vladimir Ivanisenko IB-PAS 2014 |
|
| Promedia — база данных, интегрирующая информацию о молекулярно-генетических путях, в которых участвуют летучие соединения – потенциальные биомаркеры заболеваний, имеющие значение для неинвазивной диагностики Сайк О.В., Деменков П.С., Мошкин М.П., Иванисенко В.А. 8-я Всероссийская научно-практическая конференция с международным участием «МОЛЕКУЛЯРНАЯ ДИАГНОСТИКА 2014» |
|
| HCV replication modeling: drug resistance phenomenon Иванисенко Никита Владимирович, Мищенко Елена Леонидовна, Иванисенко Владимир Александрович International Symposium on Integrative Bioinformatics 2013 |
|
| МОЛЕКУЛЯРНЫЕ МЕХАНИЗМЫ ВЗАИМОДЕЙСТВИЯ БЕЛКОВ CRMB ВИРУСА ОСПЫ КОРОВ И ВИРУСА НАТУРАЛЬНОЙ ОСПЫ С ФАКТОРОМ НЕКРОЗА ОПУХОЛЕЙ ЧЕЛОВЕКА Иванисенко Никита Владимирович, Трегубчак Т.В., Сайк О.В., Щелкунов С.Н., Колчанов Н.А., Иванисенко В.А. XXI Российский национальный конгресс "Человек и Лекарство" |
|
| Mathematical model for subgenomic Hepatitis C Virus replication: impact of drug resistance emergence on long-term kinetics of NS3 protease inhibitors action Ivanisenko N., Mishchenko E., Akberdin I., Demenkov P., Kozlov K., Todorov D., Gursky V.V., Samsonova M.G., Samsonov A.M., Clausznitzer D., Kaderali L., Kolchanov N.A., Ivanisenko V.A. 9th BGRS\SB-2014 |
|
| KINET 1.0 - a new web database on kinetics data and parameters for E.coli metabolic pathways Ermak T.V., Akberdin I.R., Timonov V.S., Mischenko E.L., Oshchepkova E.A., Perfilyeva O.A., Smirnova O.G., Khlebodarova T.M., Likhoshvai V.A. 9th BGRS\SB-2014 |
|
| Database of frame models of genetic regulation of the metabolic processes associated with diseases O.V.Saik, P.S. Demenkov, V.A. Ivanisenko Девятая Международная конференция по биоинформатике регуляции и структуры генома и системной биологии [Bioinformatics of Genome Regulation and Structure\Systems Biology — BGRS\SB-2014] |
|
| The Сompilation of Human Gene Networks Controlling Feeding Behavior and Thermoregulation E.V. Ignatieva, O.V.Saik, O.A. Podkolodnaya, N.N. Podkolodnaya, P.S. Demenkov, E.S. Tiys, E.A. Oshchepkova, E.A. Ananko, N.V. Ivanisenko, O.V. Popik, N.L. Podkolodny, V.A. Ivanisenko, D.A. Afonnikov, N.A. Kolchanov, E.I. Rogaev The first international scientific conference "Science of the Future" |
|
| Моделирование сердечно-сосудистых заболеваний с использованием индуцированных плюрипотентных стволовых клеток человека Дементьева Е.В., Григорьева Е.В., Вялкова А.В., Медведев С.П., Шевченко А.И., Елисафенко Е.А., Байрамова С.А., Покушалов Е.А., Иванисенко Н.В., Иванисенко В.А., Караськов А.М., Сухих Г.Т., Закиян С.М. Первый всероссийский симпозиум «Новейшие методы клеточных технологий в медицине» |
|
| Application of human induced pluripotent stem cells for modelling long QT syndrome Dementyeva E.V., Grigor’eva E.V., Vyalkova A.V., Medvedev S.P., Shevchenko A.I., Elisaphenko E.A., Bairamova S.A., Pokushalov E.A., Ivanisenko N.V., Ivanisenko V.A., Karaskov A.M., Sukhikh G.T., Zakian S.M. IV Международная научно-практическая конференция «Постгеномные методы анализа в биологии, лабораторной и клинической медицине» |
|
| Using ANDSystem for automated literature mining and protein interactome networks reconstruction O.V. Saik, E.S. Tiys, T.V. Ivanisenko, P.S. Demenkov, L.H. Pastushkova, I.M. Larina, E.N. Nikolaev, N.A. Kolchanov, V.A. Ivanisenko HUPO2014 the 13th World Congress of the Human Proteome Organization |
|
| MOLECULAR MECHANISMS OF INTERACTION OF ORTHOPOXVIRAL CrmB PROTEINS WITH TUMOR NECROSIS FACTOR Nikita Ivanisenko, Tatiana Tregubchak, Olga Saik, Sergey Shchelkunov, Vladimir Ivanisenko 20th European Symposium on Quantitative Structure-Activity Relationships - EuroQSAR-2014 |
|
| ANDSystem: automated literature mining and interactome networks reconstruction in the area of biology O.V. Saik, E.S. Tiys, T.V. Ivanisenko, P.S. Demenkov, N.A. Kolchanov, V.A. Ivanisenko HUPO2014 the 13th World Congress of the Human Proteome Organization |
|
| Microbial Communities of the Thermal Springs of the Geyser Valley and Uzon Caldera (Kamchatka) Using Pyrosequencing A.V. Bryanskaya, A. S. Rozanov, T. K. Malup, E. V. Lasareva, O. P. Taran, T. V. Ivanisenko, V. A. Ivanisenko, N. A. Kolchanov, S. E. Peltek 10th International Congress on Extremophiles |
|
| An integrated study to analyze salt lake microbial community structure (Novosibirsk oblast, Russia) A. Bryanskaya, A. Rozanov, T. Malup, T. Aleshina, E. Lazareva, O. Taran, T. Goryachkovskaya, V. Ivanisenko, S. Peltek 12th International Conference on Salt Lake Research |
|
| Logical modelling of NANOG-depended transcriptional gene network of embyonic carcinoma stem cells. Stepanenko I.L., Ivanisenko V.A. The Ninth International Conference on Bioinformatics of Genome Regulation and Structure \Systems Biology (BGRS'2014) |
|
| RECONSTRUCTION OF ASSOCIATIVE GENE NETWORKS SPECIFIC TO TARGET BIOLOGICAL PROCESSES AND PHENOTYPIC TRAITS E.S. Tiys, P.S. Demenkov, O.V. Saik, O.V. Popik, V.A. Ivanisenko BGRS\SB-2014 |
|
| К ПРОБЛЕМЕ ДИСТРОПИИ: ТУБЕРКУЛЕЗ И БРОНХИАЛЬНАЯ АСТМА ЕЮ Брагина, ЕС Тийс, МБ Фрейдин, ЛА Конева, ВА Иванисенко, ПС Деменков, НА Колчанов, ВП Пузырёв ГЕНЕТИКА ЧЕЛОВЕКА И ПАТОЛОГИЯ Проблемы эволюционной медицины |
|
| A machine learning analysis of urine proteomics in space-flight simulations Binder H., Wirth H., Arakelyan A., Lembcke K., Tiys E.S., Ivanishenko V., Kolchanov N.A., Kononikhin A., Popov I., Nikolaev E.N., Pastushkova L., Larina I.M BGRS\SB-2014 |
|
| Identifying overrepresented biological processes in cosmonauts on the first day Pastushkova L.H., Kononihin A.S., Tiys E.S., Obraztsova O.A., Dobrohotov I.V., Kireev K.S., Ivanisenko V.A., Nikolaev E.N., Larina I.M. BGRS\SB-2014 |
|
| THE MAMMALIAN CIRCADIAN CLOCK: COMPUTER ANALYSIS OF GENE NETWORK Подколодная О.А., Подколодная Н.Н., Подколодный Н.Л. The 9th International Conference on Bioinformatics of Genome Regulation and Structure\Systems Biology (BGRS\SB’14) |
|
| 2013 | L-MOLKERN software for capturing polarization effects in free energy calculations Fomin E.S., Alemasov N.A. VII Moscow International Congress “Biotechnology: State of the Art and Prospects of Development” |
| МЕТОД СВЯЗАННЫХ ЯЧЕЕК С СОРТИРОВКОЙ ВЗАИМОДЕЙСТВИЙ ДЛЯ ВЕКТОРИЗОВАННЫХ РАСЧЕТОВ В МОЛЕКУЛЯРНОЙ ДИНАМИКЕ Матвиенко С.А., Алемасов Н.А., Фомин Э.С. Second International Conference Cluster Computing 'CC 2013' |
|
| Computer analysis of transcription factor binding sites in genome scale based on ChIP-seq data Yuriy L. Orlov, Vladimir A. Ivanisenko, Alexey V. Kochetov, Nikolay A. Kolchanov German-Russian Forum Biotechnology, Rostock 2013 |
|
| Сравнительный анализ состава микробного сообщества воды и бентоса термального источника Заварзина, кальдера Узон, Камчатка Розанов А.С., Брянская А.В., Малуп Т.К., Лазарева Е.В., Таран О.П., Иванисенко Т.В., Иванисенко В.А., Жмодик С.М., Колчанов Н.А., Пельтек С.Е. международная научная конференция «Экология и геохимическая деятельность микроорганизмов экстремальных местообитаний» |
|
| Analysis of hepatitis C associative networks Saik O.V., Demenkov P.S., Tiys E.S., Ivanisenko V.A. Международная научная конференция "Влияние научных исследований/ Wplyw badan naukowych", Быдгощ, 2013 |
|
| Анализ принадлежности белков к функциональным группам Gene Ontology, имеющих динамику присутственности в моче космонавтов Сайк Ольга Владимировна, Тийс Евгений Сергеевич, Доброхотов Игорь Владимирович, Образцова Ольга Анатольевна, Пастушкова Людмила Ханифовна, Иванисенко Владимир Александрович Международная научно-практическая конференция "Медицина XXI века", Смоленск, 2013. |
|
| Reconstruction of squamous cell carcinoma “associome” based on analysis of differential gene expression according to RNAseq data and information stored in databases O.V. Saik, P.S. Demenkov, V.A. Ivanisenko Школа молодых ученых "Системная биология и биоинформатика", Новосибирск, 2013. |
|
| Analysis of RNAseq digital gene expression for expanding disease “associome” reconstructed based on information stored in databases Saik O.V., Demenkov P.S., Ivanisenko V.A. Международная конференция "Высокопроизводительное секвенирование в геномике", Новосибирск, 2013. |
|
| 2012 | A stochastic model for suppression of subgenomic hepatitis C virus replication in Huh-7 cells. N.V. Ivanisenko, E.L. Mishchenko, I.R. Akberdin, P.S. Demenkov, V.A. Likhoshvai, M.G. Samsonova, D. Clausznitzer, L. Kaderali, N.A. Kolchanov and V.A. Ivanisenko Международный симпозиум "Syspatho-Системная биология и медицина" |
| Protein thermal stability study using NAMD on high-performance cluster N.A. Alemasov, E.S. Fomin 8th International Conference on the Bioinformatics of Genome Regulation and Structure\Systems Biology (BGRS\SB-2012) |
|
| HIGH PERFORMANCE COMPUTING IN BIOINFORMATICS: CASE STUDIES N. L. Podkolodnyy, P.S. Demenkov, K.V. Gunbin, Y.L. Orlov, E.S. Fomin, N.A. Alemasov, F.A. Kazantsev, O.V. Vishnevsky, V.A.Ivanisenko, D.A. Afonnikov, N.V. Kuchin, B.M. Glinsky, N.A. Kolchanov The eighth international conference on bioinformatics of genome regulation and structure \ System biology (BGRS\SB'2012) |
|
| Assembly of genomes. Analysis of metagenomic data. Demenkov P.S., Ivanisenko T.V, Ivanisenko V.A. Integrative Biological Pathway Analysis and Simulation |
|
| Text- and Datamining for biological network analysis Demenkov P.S., Ivanisenko T.V., Ivanisenko V.A. Integrative Biological Pathway Analysis and Simulation |
|
| Automated literature mining for biomedicine and biotechnology Иванисенко Тимофей Владимирович German-Russian Forum Biotechnology 2012 |
|
| Datamining and biological networks P.S. Demenkov, T.V. Ivanisenko, V.A. Ivanisenko IB-PAS 2012 |
|
| Reconstruction of the associative genetic networks based on integration of automated text-mining methods and protein-ligand interaction prediction T.V. Ivanisenko, P.S. Demenkov, V.A. Ivanisenko Bioinformatics of Genome Regulation and Structure\Systems Biology — BGRS\SB-2012 |
|
| Associative network discovery system (ANDSystem): automated literature mining tool for extracting relationships between diseases, pathways, proteins, genes, microRNAs and metabolites V.A. Ivanisenko, P.S. Demenkov, T.V. Ivanisenko, E.S. Tiys Bioinformatics of Genome Regulation and Structure\Systems Biology — BGRS\SB-2012 |
|
| SUPPRESSION OF SUBGENOMIC HCV RNA BY NS3 PROTEASE ANTIVIRALS IN CELLS: A BASIC STOCHASTIC MATHEMATICAL MODEL Ivanisenko N.V., Mishchenko E.L., Akberdin I.R., Demenkov P.S., Likhoshvai V.A., Kolchanov N.A., Ivanisenko V.A. The Eighth International Conference On Bioinformatics of Genome Regulation and Structure\Systems Biology |
|
| Application of conformational peptides for analysis of allergenic proteins Брагин Анатолий Олегович, Деменков Павел Сергеевич, Иванисенко Владимир Александрович BGRS-2012 |
|
| APPLICATION OF CONFORMATIONAL PEPTIDES FOR ANALYSIS OF ALLERGENIC PROTEINS A.O. Bragin, P.S. Demenkov, V.A. Ivanisenko PTB 2012 |
|
| Promedia — база данных белков, генов, метаболитов, молекулярно-генетических путей, имеющих значение для разработки средств диагностики и лечения заболеваний Сайк Ольга Владимировна, Деменков Павел Сергеевич, Иванисенко Владимир Александрович II Российский конгресс с международным участием «Молекулярные основы клинической медицины – возможное и реальное» |
|
| PATTERNS OF MIRNA BINDING SITES LOCATION IN 3`UTRs OF HUMAN TRANSCRIPTS D.N. Shtokalo, O.V. Saik, G.St.Laurent III, A.Kel BGRS/SB’2012, Novosibirsk |
|
| Distributed RESTFUL-WEB-services for the recognition and analysis of gene networks. Podkolodnyy N.L., Semenychev A.V., Borovsky V.G., Rasskazov D.A., Ananko E.A., Ignatieva E.V., Podkolodnaya N.N. Podkolodnaya O.A. The Eighth International Conference on Bioinformatics of Genome Regulation and Structure\Systems Biology (BGRS'2012) |
|
| Dynamical and structural analysis of an apoptosis network in hepatitis C. Stepanenko I.L., Smirnova O.G. Eghth International Conference on Bioinformatics of Genome Regulation and Structure \Systems Biology (BGRS'2012) |
|
| Application of the ANDVisio computer system to the interpretation of biological functions of proteins, differentially expressed in bronchoalveolar lavage of mice after a one-time intranasal administration of SiO2 nanoparticles. E.V. Ignatieva, V.A. Ivanisenko, E.S. Tiys, P.S. Demenkov, M.P. Moshkin, S.E. Peltek. The Eighth International Conference on Bioinformatics of Genome Regulation and Structure \ Systems Biology (BGRS/SB'12) |
|
| A central regulatory circuit of arabidopsis circadian clock gene network Smirnova O.G., Stepanenko I.L. Eghth International Conference on Bioinformatics of Genome Regulation and Structure \Systems Biology (BGRS'2012) |
|
| Исследование закономерностей развития самоорганизующихся социально-биологических систем Подколодный Н.Л., Деменков П.С., Иванисенко В.А., Лашин С.А., Семенычев А.В., Рассказов Д.А., Титов И.И., Колчанов Н.А. XIV Российская конференция с международным участием "Распределенные информационные и вычислительные ресурсы" (DICR-2012) |
|
| Распределенная система RESTful-web-сервисов для реконструкции и анализа генных сетей Подколодный Н.Л., Семенычев А.В., Рассказов Д.А., Деменков П.С., Боровский В.Г., Подколодная Н.Н., Ананько Е.А., Игнатьева Е.В., Подколодная О.А. XIV Российская конференция с международным участием "Распределенные информационные и вычислительные ресурсы" (DICR-2012) |
|
| Разработка новых средств для аллерген-специфической иммунотерапии на основе дизайна конформационных пептидов Брагин Анатолий Олегович Тийс Евгений Сергеевич «Участник молодежного научно-инновационного конкурса» («УМНИК») Медицина Будущего |
|
| INFLUENCES OF PROTEIN FUNCTIONAL SITES ENCODING I.V. Medvedeva, P.S. Demenkov, Ivanisenko V. A. BGRS-2012 |
|
| 2011 | Компьютерная протеомика: от метагенома к метопротеому Иванисенко В.А., Деменков П.С., Пинтус С.С. Рабочее совещание "Высокопроизводительные вычисления в биоинформатике" |
| ITMSys: Interactive Text-mining System Ivanisenko T.V. International German/Russian Summer School on Integrative Biological Pathway Analysis and Simulation 2011 |
|
| Analysis of protein functional sites encoding features Медведева И.В., Деменков П.С., Иванисенко В.А. International German/Russian Summer School on Integrative Biological Pathway Analysis and Simulation |
|
| Analysis of protein functional sites encoding features Медведева Ирина Вадимовна, Деменков Павел Сергеевич, Иванисенко Владимир Александрович ISMB/ECCB 2011 |
|
| Моделирование генных сетей: система GeneNet в 2011 году. Тимонов В.С., Гунбин К.В., Казанцев Ф.В., Акбердин И.Р., Деменков П.С., Лашин С.А., Ананько Е.А., Иванисенко В.А., Подколодный Н.Л., Колчанов Н.А. Международная конференция "Современные проблемы математики, информатики и биоинформатики" |
|
| The visual reconstruction and analysis of biomolecular systems: The GeneNet software in 2011 Vladimir Timonov, Fedor Kazantsev, Konstantin Gunbin, Ilya Akberdin, Pavel Demenkov, Elena Ananko, Nikolay Podkolodny, Vladimir Ivanisenko, Nikolay Kolchanov International German/Russian Summer School on Integrative Biological Pathway Analysis and Simulation |
|
| A mathematical model for the suppression of subgenomic Hepatitis C virus replication in Huh-7 cells in the presence of the NS3 protease inhibitors Мищенко Е., Иванисенко Н., Акбердин И., Лихошвай В., Козлов К., Гурский В., Самсонова М., Колчанов Н., Иванисенко В. International Conference on Systems Biology (ICSB 2011), Хайдельберг, Германия, 28 августа-1 сентября |
|
| Математическая модель подавления репликации субгеномного репликона вируса гепатита С в клетке с ингибитором NS3 протеазы SCH503034 Мищенко Е., Иванисенко Н., Акбердин И., Лихошвай В., Козлов К., Гурский В., Самсонова М., Колчанов Н., Иванисенко В. Санкт-Петербургский научный форум “Наука и общество. Физиология и медицина 21 века. VI Петербургская встреча лауреатов нобелевской премии”, Санкт-Петербург, Россия, 19-23 сентября |
|
| Biological literature mining. Application of associative network discovery system in systems biology Demenkov P.S., Ivanisenko T.V., Ivanisenko V.A. International German/Russian Summer School on Integrative Biological Pathway Analysis and Simulation |
|
| Улучшенные алгоритмы расчёта свободной энергии для определения термостабильности белковых комплексов Алемасов Н.А., Фомин Э.С. II международная научно-практическая конференция «Постгеномные методы анализа в биологии, лабораторной и клинической медицине: геномика, протеомика, биоинформатика» |
|
| Программный пакет GENENETSTUDIO: визуальное моделирование сетевых моделей биомолекулярных систем. Тимонов В.С., Гунбин К.В., Казанцев Ф.В., Акбердин И.Р., Деменков П.С., Подколодный Н.Л. Третья школа молодых ученых «Биоинформатика и системная биология» |
|
| A gene regulatory network model for vernalization and seasonal flowering response in winter wheat. Stepanenko I.L., Smirnova O.G., Titov I.I International Conference Wheat Resources and Genomics (WGRG 2011) |
|
| Компьютерный анализ связи конформационных пептидов с аллергенностью белков Брагин Анатолий Олегович, Деменков Павел Сергеевич, Иванисенко Владимир Александрович Постгеномные методы анализа в биологии, лабораторной и клинической медицине: геномика, протеомика, биоинфоратика, г. Новосибирск, 14-17 ноября 2011 г. |
|
| Компьютерный анализ связи конформационных пептидов с аллергенностью белков. Брагин Анатолий Олегович, Деменков Павел Сергеевич, Иванисенко Владимир Александрович Третья школа молодых ученых “Биоинформатика и системная биология” |
|
| VISUALIZATION AND ANALYSIS OF A CARDIO VASCULAR DISEASE-RELATED BIOLOGICAL NETWORK COMBINING TEXT MINING AND DATA WAREHOUSE APPROACHES Ralf Hofestädt, Björn Sommer, Evgeny Tiys, Benjamin Kormeier, Klaus Hippe, Sebastian Janowski, Timofey Ivanisenko, Anatoly Bragin, Patrizio Arrigo, Pavel Demenkov, Alexey Kochetov, Vladimir Ivanisenko, Nikolay Kolchanov Moscow Conference on Computational Molecular Biology 2011 |
|
| Исследование масштабируемости алгоритмов поиска ближайших соседей в методе молекулярной динамики Матвиенко С.А., Алемасов Н.А., Фомин Э.С. II международная научно практическая конференция Постгеномные методы анализа в биологии, лабораторной и клинической медицине: геномика, протеомика, биоинформатика |
|
| Автоматическая реконструкция ассоциативных генных сетей на примере анализа протеома мочи Тийс Е.С., Деменков П.С., Доброхотов И.В., Валеева О.А., Пастушкова Л.Х., Николаев Е.Н., Ларина И.М., Колчанов Н.А., Иванисенко В.А. Постгеномные методы анализа в биологии, лабораторной и клинической медицине: геномика, протеомика, биоинфоратика, г. Новосибирск, 14-17 ноября 2011 г. |
|
| Метагеном озера «Кротовья ляга» Иванисенко В.А., Деменков П.С., Иванисенко Т.В., Розанов А.С., Брянская А.В., Кострюкова Е.С., Левицкий С.А., Селезнева О.В., Чукин М.М., Ларин А.К., Кондратов И.Г., Лазарев В.Н., Пельтек С.Е., Говорун В.М., Колчанов Н.А. Постгеномные методы анализа в биологии, лабораторной и клинической медицине |
|
| Анализ текстов (Text-mining) и ассоциативные сети в области молекулярной биологии Деменков П.С., Иванисенко Т.В., Иванисенко В.А. Постгеномные методы анализа в биологии, лабораторной и клинической медицине |
|
| Создание базы данных летучих соединений, потенциальных маркеров заболеваний Сайк Ольга Владимировна, Иванисенко Владимир Александрович, Деменков Павел Сергеевич Interra, секция биомедицина, Новосибирск, 2011 |
|
| PROMEDIA – база данных химических соединений потенциальных биомаркеров заболеваний Сайк Ольга Владимировна, Иванисенко Владимир Александрович, Деменков Павел Сергеевич Interra, секция биоинформатика и системная биология, Новосибирск, 2011 |
|
| Database of metabolites, enzymes, transcription factors associated with diseases Olga Saik, Vladimir Ivanisenko, Pavel Demenkov RCIBC, Новосибирск, 2011 |
|
| 2010 | Microbial metagenome annotation with bioinformatics and computational systems biology methods Ivanisenko, Demenkov, Pintus, Ivanisenko, Goncharova, Kostjukova, Levitski, Selezneva, .... Kolchanov 7th International conference on bioinformatics of genome regulation and structure\system biology (BGRS/SB'20010). June 20-27, 2010, Novosibirsk |
| Modeling of the suppressive effect of NS3 protease inhibitor on HCV subgenomic replicon replication in Huh-7 cells Bezmaternykh K.D., Mishchenko E.L.,Likhoshvai V.A., Ivanisenko V.A., Kolchanov N.A. 7th International conference on bioinformatics of genome regulation and structure\system biology (BGRS/SB'20010). June 20-27, 2010, Novosibirsk |
|
| Reconstruction of molecular genetic networks: text mining as a tool for knowledge discovery V.A. Ivanisenko, P.S. Demenkov, T.V. Ivanisenko Joint Indo-Russian Workshop "Predictive Biology using Systems and Integrative analysis and Methods", November 15-19, 2010, p 8-9, Chandigarh, India |
|
| Computer Sytem SitEx for analyzing protein functional sites in eukaryotic gene structure Медведева И. В., Деменков П. С., Иванисенко В. А. The Young Scientists School "Bioinformatics and system biology", Novosibirsk, Russia, 28-29 June 2010 |
|
| Разработка базы данных химических соединений - потенциальных маркеров заболеваний Иванисенко В.А., Сайк О.В., Деменков П.С., Мошкин П.М. XVII Российский национальный конгресс "Человек и лекарство"., Москва, 16 апреля 2010 г. |
|
| Генная сеть активации макрофага: участие ксенобиотков в регуляции экспрессии генов цитокинов Ощепкова Е.А., Ощепков Д.Ю., Кашина Е.В., Антонцева Е.В., Фурман Д.П., Мордвинов В.А. Всероссийская научная конференция "Молекулярно-генетические основы функционирования цитокиновой сети в норме и при патологии", 15-17 сентября 2010г. |
|
| Интегрированная система для информационной поддержки исследования механизмов регуляции транскрипции Н.Л. Подколодный, Е.В. Игнатьева, Д.А. Рассказов, О.А. Подколодная, Е.А. Ананько, Н.Н. Подколодная, Е.М. Залевский Всероссийская научная конференция "Электронные библиотеки: перспективные методы и технологии, электронные коллекции" – RCDL’2010, Казань, Россия, 2010 |
|
| Сравнительный анализ производительности процессоров PowerXCell8i и Intel X7560 на примере задач молекулярной динамики Фомин Э.С., Н.А. Алемасов, С.А.Матвиенко Высокопроизводительные параллельные вычисления на кластерных системах HPC2010, Материалы X Международной конференции, г.Пермь, 1-3 ноября, 2010 г. |
|
| Математическое моделирвание репликации репликона вируса гепатита С: предсказание эффетивности действия новых потенциальных лекарств Мищенко Е.Л., Иванисенко Н.В., Мищенко А.М., Лихошвай В.А., Иванисенко В.А. Международная научно-практическая конференция "Высокие технологии, фундаментальные и прикладные исследования в физиологии и медицине" 23-26 ноября, 2010, г. Санкт-Петербург |
|
| Экспериментально-компьютерное исследование генов, вовлекаемых в реакцию макрофага на диоксин Ощепков Д.Ю., Ощепкова Е.А., Кашина Е.В., Антонцева Е.В., Фурман Д.П., Мордвинов В.А. Международная научно-практическая конференция "Высокие технологии, фундаментальные и прикладные исследования в физиологии и медицине" 23-26 ноября, 2010, г. Санкт-Петербург |
|
| Компьютерный анализ метагеномных данных: предсказание количественной величины специфической активности белков Иванисенко В.А., Деменков П.С., Пинтус С.С., Иванисенко Т.В., Гончарова Н.И., Розанов А., Брянская А., Кострюкова Е.С., Левитский С.А., Селезнева О.В., Чукин М.М., Ларин А.К., Кондратов И.Г., Лазарев В.Н., Пельтек С.Е., Говорун В.М. Постгеномные методы анализа в биологии, лабораторной и клинической медицине |
|
| Computer analysis of conformationalpeptides in protein families A.O. Bragin, P.S. Demenkov, V.A. Ivanisenko 7th International conference on bioinformatics of genome regulation and structure\system biology (BGRS/SB'20010). June 20-27, 2010, Novosibirsk |
|
| Analisys of human proteome based on protein stability to mutations P.S. Demenkov, O.A. Korepanova, T.V. Ivanisenko, V.A. Ivanisenko 7th International conference on bioinformatics of genome regulation and structure\system biology (BGRS/SB'20010). June 20-27, 2010, Novosibirsk |
|
| ITMSys: An interactive web-based text-mining system for automated analysis of the full-text articles Ivanisenko T.V., Demenkov P.S., Ivanisenko N.V., Ivanisenko V.A. 7th International conference on bioinformatics of genome regulation and structure\system biology (BGRS/SB'20010). June 20-27, 2010, Novosibirsk |
|
| ANDCell and ANDNanobiotech: Associative network discovery systems in systems biology and nanobiotechnology V.A. Ivanisenko, P.S. Demenkov, T.V. Ivanisenko, N.L. Podkolodny 7th International conference on bioinformatics of genome regulation and structure\system biology (BGRS/SB'20010). June 20-27, 2010, Novosibirsk |
|
| Computer system SitEx for analyzing protein functional sites in eukaryotic gene structure I.V. Medvedeva, P.S. Demenkov, V.A. Ivanisenko 7th International conference on bioinformatics of genome regulation and structure\system biology (BGRS/SB'20010). June 20-27, 2010, Novosibirsk |
|
| The nanobiotechnology database V.A. Ivanisenko, N.L. Podkolodnyy, P.S. Demenkov, T.V. Ivanisenko, N.N. Podkolodnaya, T.M. Khlebodarova, E.V. Ignatieva, O.A. Podkolodnaya, E.A. Ananko, and N.A. Kolchanov 7th International conference on bioinformatics of genome regulation and structure\system biology (BGRS/SB'2010). June 20-27, 2010, Novosibirsk |
|
| A computer analysis of functional relationships between genes associated with multifactorial diseases: pre-eclampsia as an example E. Tiys, P. Demenkov, E. Vashukova, A. Glotov, V. Ivanisenko 7th International conference on bioinformatics of genome regulation and structure\system biology (BGRS/SB'20010). June 20-27, 2010, Novosibirsk |
|
| Promoters of the genes encoding the macrophage transcription factors contain binding sites for aryl hydrocarbon receptor. Oshchepkova E.A., Oshchepkov D.Y., Kashina E.V., Antontseva E.V., Shamanina M.Y., Mordvinov V.A., Furman D.P. Seventh international conference on bioinformatics of genome regulation and structure\system biology |
|
| Detection of dioxin responsive elements in the promoter regions of the genes encoding macrophage cytokines. Oshchepkova E.A., Oshchepkov D.Y., Kashina E.V., Antontseva E.V., Furman D.P., Mordvinov V.A. Seventh International Conference on Bioinformatics of Genome Regulation and Structure and Systems Biology (BGRS_SB’10),Новосибирск, Россия, 20-27 июня |
|
| Detection of dioxin responsive elements in the promoter regions of the macrophage transcription factors and cytokines genes. Oshchepkova E.A., Oshchepkov D.Y., Kashina E.V., Antontseva E.V., Shamanina M.Y., Mordvinov V.A., Furman D.P. “From Functional Genomics to Systems Biology”, |
|
| Компьютерно-экспериментальное изучение сайтов связывания транскрипционных факторов. Ощепков Д.Ю., Климова Н.В., Антонцева Е.В., Левицкий В.Г., Ощепкова Е.А., Кашина Е.В., Игнатьева Е.В., Васильев Г.В., Пономаренко М.П., Драчкова И.А., Савинкова Л.К., Фурман Д.П., Мордвинов В.А., Меркулова Т.И. I международной научно-практической конференции «Постгеномные методы анализа в биологии, лабораторной и клинической медицине» |
|
| Экспериментально-компьютерное исследование генов, вовлекаемых в реакцию макрофага на диоксин. Ощепков Д.Ю., Ощепкова Е.А., Кашина Е.В., Антонцева Е.В., Фурман Д.П., Мордвинов В.А. Международная научно-практическая конференция «Высокие технологии, фундаментальные и прикладные исследования в физиологии и медицине» |
|
| Testing of functional activity of putative dioxin responsive elements in promoter regions of genes, encoding macrophageal transcription factors and cytokines Kashina E.V., Antontseva E.V., Katokhin A.V., Oshchepkova E.A., Oshchepkov D.Y., Shamanina M.Y., Grishanova A.Y., Furman D.P, Mordvinov V.A. The seventh international conference on bioinformatics of genome regulation and structure\system biology |
|
| БАЗА ДАННЫХ ХИМИЧЕСКИХ СОЕДИНЕНИЙ, ПОТЕНЦИАЛЬНЫХ БИОМАРКЕРОВ ЗАБОЛЕВАНИЙ О.В. Сайк, П.С. Деменков, Т.В. Иванисенко, М.П. Мошкин, В.А. Иванисенко Десятая всероссийская научно-практическая конференция «Интеллектуальный потенциал ученых России» |
|
| БАЗА ДАННЫХ ХИМИЧЕСКИХ СОЕДИНЕНИЙ, ИМЕЮЩИХ ПОТЕНЦИАЛЬНОЕ ЗНАЧЕНИЕ ДЛЯ НЕИНВАЗИВНОЙ ДИАГНОСТИКИ ЗАБОЛЕВАНИЙ О.В. Сайк, П.С. Деменков, Т.В. Иванисенко, М.П. Мошкин, В.А. Иванисенко II Всероссийская научная конференция с международным участием "Научное творчество ХХI века" |
|
| Computer system for analysis of mechanisms of transcription regulation in eukaryotes Podkolodnyy N.L., Ignatieva E.V., Rasskazov D.A., Ananko E.A., Podkolodnaya O.A., Podkolodnaya N.N., Zalevsky E.M. 7th International conference on bioinformatics of genome regulation and structure\system biology (BGRS/SB'20010). June 20-27, 2010, Novosibirsk |
|
| Computer analysis of stress response network E.coli. Stepanenko I.L., Titov I.I. Seventh International Conference on Bioinformatics of Genome Regulation and Structure \Systems Biology (BGRS'2010) |
|
| Computer analysis of conformationalpeptides in protein families A.O. Bragin, P.S. Demenkov, V.A. Ivanisenko The 7th International Conference on Bioinformatics of Genome Regulation and Structure |
|
| Computer analysis of conformational peptides in protein families Bragin A.O., Demenkov P.S., Ivanisenko V.A summer school and workshop Integrative Biological Pathway Analysis and Simulation – 2010 |
|
| 2009 | модификация фермента ксилозоизомеразы E.Coli дисульфидным мостиком для повышения стабильности Розанов А.С., Цырульников А.О., Иванисенко В.А., Пельтек С.Е. 13 - международная школа конференция молодых ученых, 28 сентября - 2 октября. Биология наука 21-го века |
| Associative network discovery systems in systems biology and nanobiotechnology Ivanisenko V.A. International Autumn School for Young Scientists on Computational Systems Biology and Bioinformatics |
|
| Suppressive effect of potencial individual drugs or their combinations on HCV replication in cells: a mathematical model Mishchenko E.L. International Autumn School for Young Scientists on Computational Systems Biology and Bioinformatics |
|
| Автоматическая экстракция знаний из научных публикаций в области системной биологии и нанобиотехнологии Иванисенко В.А., Деменков П.С., Иванисенко Т.В., Подколодный Н.Л., Колчанов Н.А. IV Петербургская встреча Нобелевских лауреатов. С.-Петербург. 21-25 сентября 2009г |
|
| ANDCell and ANDNanobiotech: associative network discovery systems in systems biology and nanobiotechnology Vladimir A. Ivanisenko, P.S. Demenkov, T.V. Ivanisenko Proceedings of the German-Russian Forum Biotechnology GRFB’09, 2009 |
|
| Использование архитектуры Cell/B.E. для ускорения выполнения пакета молекулярного моделирования MOLKERN Алемасов Н.А. XVI Международная конференция студентов, аспирантов и молодых ученых "Ломоносов", Москва, 14-17 апреля 2009 |
|
| Алгоритм расчёта ближних невалентных взаимодействий на процессоре PowerXCell8i. SPE-центричная модель Алемасов Н.А., Фомин Э.С. Девятая международная конференция-семинар "Высокопроизводительные параллельные вычисления на кластерных системах", Владимирский государственный университет им. А.Г. и Н.Г. Столетовых, Владимир, 2–3 ноября 2009 года |
|
| Нанобиотехнология: извлечение знаний методами text-mining Колчанов Н.А., Иванисенко В.А., Деменков П.С., Подколодный Н.Л. Конференция "Химическая биология – Фундаментальные проблемы бионанотехнологии", 10 – 14 июня 2009 г., Новосибирск |
|
| Изучение влияния на термостабильность ксилозоизомераза e.coli введения межъсубеденичного дисульфидного мостика Розанов А.С., Цырульников А.О., Иванисенко В.А., Пельтек С.Е. Конференция IV Международная конференция "Биология: от молекулы до биосферы" 17 - 21 ноября была перенесена |
|
| Информационная система ANDCell-ANDVisio для построения ассоциативных сетей в области молекулярной биологии Деменков П. С., Яркова Е. Э., Иванисенко В. А. Седьмая международная конференция памяти академика А.П. Ершова. Рабочий семинар "Наукоемкое программное обеспечение" 15-19 июня 2009. Новосибирск |
|
| Нанобиотехнология: извлечение знаний методами text-mining Иванисенко В.А., Деменков П. С., Подколодный Н.Л., Яркова Е. Э., Иванисенко Т. В., Хлебодарова Т.М., Колчанов Н.А. Пятый московский международный конгресс “Биотехнология: состояние и перспективы развития” |
|
| 2008 | Data mining and network construction Ivanisenko V.A. 11th Meeting of the German/Russian Virtual Network of Bioinformatics for "Computational Systems Biology". August 25-26, 2008. Bielefeld, Germany |
| A mathematical model for the suppressive effect of subgenomic hepatitis c virus replication in cells in the presence of potential individual drugs or their combinations E.L. Mishchenko, K.D. Bezmaternykh, V.A. Likhoshvai, V.A. Ivanisenko, N.A. Kolchanov. 6th international conference on bioinformatics of genome regulation and structure BGRS’2008, Novosibirsk, June 22-28 |
|
| Andcell: a computer system for automated extraction of knowledge about molecular genetic interactions and regulations from pubmed abstracts and their representation as semantic association networks Vladimir A. Ivanisenko, P.S. Demenkov, T.V. Ivanisenko 6th international conference on bioinformatics of genome regulation and structure BGRS’2008, Novosibirsk, June 22-28 |
|
| Coevolution of protein domains of P53 and MDM2 – key proteins of apoptosis S.S. Pintus, V.A. Ivanisenko 6th international conference on bioinformatics of genome regulation and structure BGRS’2008, Novosibirsk, June 22-28 |
|
| Estimation of minimal drug treatment duration for clearance of an Huh-7 cell from hepatitis C virus replicon based on mathematical modeling Mishchenko E.L., Bezmaternykh K.D., Likhoshvai V.A., Ivanisenko V.A., Kolchanov N.A. 6th international conference on bioinformatics of genome regulation and structure BGRS’2008, Novosibirsk, June 22-28 |
|
| Modeling of drug effects on hepatitis C virus replication in an Huh-7 cell Mishchenko E.L., Bezmaternykh K.D., Likhoshvai V.A., Ivanisenko V.A., Kolchanov N.A. 6th international conference on bioinformatics of genome regulation and structure BGRS’2008, Novosibirsk, June 22-28 |
|
| Nanobiotechnology and its application to biomedicine: text-mining knowledge extraction and integration Ivavisenko V.A. Demenkov P.S., Yarkova E.E., Ivavisenko N.V., Ivavisenko T.V. Sournina N.Yu., Podkolodny N.L., Khlebodarova T.M., Ibragimova S.S., Smirnova O.G. 6th international conference on bioinformatics of genome regulation and structure BGRS’2008, Novosibirsk, June 22-28 |
|
| PDBSite database and PDBSiteScan tool: template-based docking and recognition of functional sites in protein 3d structure Ivanisenko V.A., Ivanisenko N.V., Ivanisenko T.V. 6th international conference on bioinformatics of genome regulation and structure BGRS’2008, Novosibirsk, June 22-28 |
|
| Protein connectivity in molecular-genetic networks depends on protein susceptibility to single mutations Demenkov P.S., Yarkova E.E., Ivanisenko V.A., Ivanisenko T.V. 6th international conference on bioinformatics of genome regulation and structure BGRS’2008, Novosibirsk, June 22-28 |
|
| Protein Functional Sites Projection on Exon Structure Of Gene for ATP-, ADP-, AMP-binding Sites I.V. Medvedeva, V.A. Ivanisenko International Autumn School for Young Scientists on Computational Systems Biology and Bioinformatics |
|
| Protein Functional Sites Ivanisenko V.A. International Autumn School for Young Scientists on Computational Systems Biology and Bioinformatics |
|
| Эффект потенциальных лекарственных препаратов на репликацию субгеномного репликона вируса гепатита C в клетке: математическая модель Е.Л. Мищенко, К.Д.Безматерных, В.А. Лихошвай, В.А. Иванисенко IV Международная конференция "Фундаментальные науки - медицине", г. Новосибирск, 2008 г. |
|
| Математическое моделирование репликации репликона ВГС в клетке. Исследование возможных клеточных факторов Безматерных К.Д., Мищенко Е.Л., Иванисенко В.А., Лихошвай В.А IV съезд микробиологов Узбекистана, тезисы докладов, г. Ташкент, 9-10 октября 2008 |
|
| Информационные ресурсы для дизайна новых геносенсоров. разработка полифункциональных геносенсоров на основе промоторов генов yfiA и dps Escherichia coli Т. М. Хлебодарова, А.В. Качко, Н.В. Тикунова, В.А. Лихошвай, Т.Ю.Степанова, Д.Ю. Ощепков, И.Л. Степаненко, Ю.Г. Матушкин IV съезд Российского Общества биохимиков и молекулярных биологов, 10-15 мая, Новосибирск, 2008 |
|
| Drug targets discovery with text-mining and bioinformatics approaches: ANDCell and PDBSiteScan tools Ivanisenko V.A., Demenkov P.S., Yarkova E.E. Joint Indo-Russian Workshop "Systems Biology and Genome Informatics of M. tuberculosis and other infectious diseases" October 12-14, 2008. Novosibirsk |
|
| ANDCell: a tool for automated knowledge extraction from PubMed and reconstruction of association networks Ivanisenko V.A., Yarkova E.E., Demenkov P.S., Kochetov A.V. Proceedings of the 5th International Symposium on Integrative Bioinformatics. P 327, 20-22 August 2008, Germany |
|
| Knowledge discovery for biotechnology, biomedicine and nanobioengineering using text- and data-mining Ivanisenko V.A., Demenkov P.S., Yarkova E.E. Russian-Polich Symposium "Perspectives of post-genomic biotechnology" in the frames of jubilee celebrations on 50 –anniversary of cooperation between the Russian and Polish Academies of Sciences, October 15-17, 2008, Moscow |
|
| Параллельная реализация программного комплекса молекулярного моделирования "MOLKERN" с использованием OpenMP Алемасов Николай Александрович Технологии Microsoft в теории и практике программирования, Конференция-конкурс работ студентов, аспирантов и молодых ученых, Новосибирск, Академгородок |
|
| Protein connectivity in molecular-genetic networks depends on protein susceptibility to single mutations Demenkov P.S., Yarkova E.E., Ivanisenko T.V., Ivanisenko V.A. 6th international conference on bioinformatics of genome regulation and structure BGRS’2008, Novosibirsk, June 22-28 |
|
| OpenMP+MPI parallel implementation of the «MOLKERN» molecular modelling software package Алемасов Н.А., Фомин Э.С. 6th international conference on bioinformatics of genome regulation and structure BGRS’2008, Novosibirsk, June 22-28 |
|
| Quantum-chemical analysis of Zn2+ binding in wild-type and G245C mutant of p53 protein. Bugakov I.V., Fomin E.S., Ivanisenko V.A. 6th international conference on bioinformatics of genome regulation and structure BGRS’2008, Novosibirsk, June 22-28 |
|
| Detection of new potentially active DRE sites in regulatory region of human genes encoding components of Ah receptor cytosolic complex. M.Y. Shamanina, E.A. Oshchepkova, D.Y. Oshchepkov, A.V. Katokhin, D.P. Furman, I.B. Tsyrlov, V.A. Mordvinov Sixth International Conference On Bioinformatics of Genome Regulation and Structure |
|
| Distribution of active site structural analogs in enzyme 3D-structures: computer analysis E.S.Teeys, V.A. Ivanisenko Sixth International Conference On Bioinformatics of Genome Regulation and Structure |
|
| SITEGA method application for genome wide prediction of p53 binding sites. Stepanenko I.L., Levitsky V.G.. The Sixth International Conference on Bioinformatics of Genome Regulation and Structure |
|
| Hepatitis C virus life cycle: mathematical models and prospect for effective therapy Mishchenko E.L. International Autumn School for Young Scientist on Computational Systems Biology and Bioinformatics |
|
| 2007 | Analysis of Distribution of Protein Functional Sites on Gene Structure Medvedeva I. V., Demenkov P. S., Ivanisenko V. A., and Kolchanov N. A. 2007 International Conference on Bioinformatics and Computational Biology (BIOCOMP'07: June 25-28, 2007) |
| Associative network and protein structure discovery: a software complex for facilitating search of targets for drugs, drug design, and evaluation of molecular toxicity Ivanisenko V.A., Demenkov P.S., Aman E.E., S.S. Pintus S.S., Kolchanov N.A. 3-rd International Conference "Basic Science for Medicine", September 2-8, 2007, Novosibirsk, Russia |
|
| Signals influencing general translational efficiency of eukaryotic mRNAs Ivanisenko VA, Mischenko EL, Kochetov A.V., Ivanisenko V.A., Titov I.I., Kolchanov N.A., Sarai A. 3-rd Moscow Conference on Computational Molecular Biology "MCCMB' 2007". July 27-31, 2007, Moscow, Russia |
|
| Associative network discovery (AND) – Software package for automated reconstruction of molecular-genetic association networks Aman E.E., Demenkov P.S., Nemiatov A., Ivanisenko V.A. 3-rd Moscow Conference on Computational Molecular Biology "MCCMB' 2007". July 27-31, 2007, Moscow, Russia |
|
| Prediction in changes of protein thermodynamic stability upon single mutations P.S. Demenkov 8th Meeting German / Russian Virtual Network on Computational Systems Biology 5th and 6th November 2007 |
|
| Математическая модель репликации РНК репликона вируса гепатита С в клетке. Построение и варианты использования Безматерных К.Д., Мищенко Е.Л., Иванисенко В.А., Лихошвай В.А., Колчанов Н.А. III Международная конференция "Фундаментальные науки - медицине", г. Новосибирск, 2-8 сентября 2007 г. |
|
| Mathematical Modeling of the HCV Drugs Combinations Effect Bezmaternykh K.D., Mishchenko E.L., Ivanisenko V.A., Likhoshvai V.A. In Proceedings of the 3-rd Moscow Conference on Computational Molecular Biology. Moscow, Russia, July 27-31, 2007 |
|
| Bioinformatics and its application in addressing the chalanges of biotechnology and medicine Ivanisenko V.A. International Conference "Scenarios for a co-ordinated approach to sustainable S&T co-operation with the eastern neighbours of the EU", Moscow 3-4 December, 2007 |
|
| Параллельная реализация задач молекулярного докинга и виртуального скрининга Алемасов Николай Александрович, Фомин Эдуард Станиславович IV Российская-германская школа по параллельным вычислениям на высокопроизводительных вычислительных системах, Новосибирск |
|
| Structure discovery – computer tools for protein analysis and search of drug target V.A. Ivanisenko, P.S. Demenkov, E.E. Aman, S.S. Pintus, E.S. Fomin. The Fifth Moscow International Congress "Biotechnology: State of the Art and Prospects of Development" 14-18 марта 2007 года |
|
| Textomics: the instrument for biological knowledge discovery E.E. Aman, P.S. Demenkov, V.A. Ivanisenko The Fifth Moscow International Congress "Biotechnology: State of the Art and Prospects of Development" 14-18 марта 2007 года |
|
| Associative Network Discovery (AND) – компьютерная система для автоматической реконструкции ассоциативных сетей молекулярно-генетических взаимодействий Aman E.E., Demenkov P.S., Nemiatov A., Ivanisenko V.A. VI Всероссийской научно – практической конференции AS’2007 (СИСТЕМЫ АВТОМАТИЗАЦИИ в образовании, науке и производстве) |
|
| Анализ картирования функциональных сайтов белков на экзонной структуре гена Медведева И. В. XLV международная научная студенческая конференция: Студент и научно-технический прогресс, Новосибирск 10-12 апреля 2007г. |
|
| Математическая модель репликации репликона вируса гепатита С в клетках Huh-7 Безматерных К.Д., Мищенко Е.Л., Иванисенко В.А., Лихошвай В.А. XLV международная научная студенческая конференция: Студент и научно-технический прогресс, Новосибирск 10-12 апреля 2007г. |
|
| Математическое моделирование комбинационного действия лекарственных препаратов против вируса гепатита С. Безматерных К.Д., Мищенко Е.Л., Иванисенко В.А., Лихошвай В.А. Международная молодежная научно-методическая конференция "Проблемы молекулярной и клеточной биологии", г. Томск |
|
| MOLKERN as new effective engine for drug discovery software Fomin E.S., Alemasov N.A., Chirtsov A.S., Fomin A.E. 4th International Symposium Computational Methods in Toxicology and Pharmacology Integrating Internet Resources (CMTPI-2007) |
|
| Detection of new potentially active DRE sites in regulatory region of human genes encoding components of Ah receptor cytosolic complex. Nedosekina EA, Oshchepkov D.Y., Katokhin AV, Kuznetsova TN, Shamanina MY, Mordvinov VA, Tsyrlov I. 27th International Symposium on Halogenated Persistent Organic Pollutants “Dioxin 2007” |
|
| 2006 | Bioinformatics and mechanisms of molecular pathology N.A. Kolchanov, V. Ivanisenko, M. Ponomarenko, E. Aman, E. Ignatieva, T. Kuznetsova, V. Mordvinov, P. Demenkov, A. Ratushny, V.Likhoshvai, E. Ananko, N. Podkolodny, N. Podkolodnaya, A.. Romaschenko 3rd International conference "Genomics, Proteomics, Bioinformatics and Nanotechnologies for Medicine" July 12-16, 2006, Novosibirsk, Russia |
| Hepatitis C virus: application of gene networks technology to searching for perspective pharmacological targets Ivanisenko VA, Mischenko EL, Bezmaternikh KD, Ratushny AV, Likhoshvai VA,Titov II, Korotkov RO, Khlebodarova TM, Kolchanov NA. 3rd International conference "Genomics, Proteomics, Bioinformatics and Nanotechnologies for Medicine" July 12-16, 2006, Novosibirsk, Russia |
|
| Phylogenetic analysis of the p53 and p63/p73 gene families Pintus SS, Ivanisenko VA 3rd International conference "Genomics, Proteomics, Bioinformatics and Nanotechnologies for Medicine" July 12-16, 2006, Novosibirsk, Russia |
|
| New Zn2+ binding sites in the p53 mutants are promising targets for antitumor drug design Fomin E.S., Oshurkov I.S., Ivanisenko V.A. 3rd International conference "Genomics, Proteomics, Bioinformatics and Nanotechnologies for Medicine" July 12-16, 2006, Novosibirsk, Russia |
|
| Analysis of the tertiary structure of the PPAR and RXR transcriptional factors and their mutant variants Aman E.E., Demenkov P.S., Ivanisenko V.A. 5th International Conference on Bioinformatics of Genome Regulation and Structure (BGRS'2006), Novosibirsk, Russia (July 14-22) |
|
| Database GenSensor as informational source for design of biosensors. Experimental development of biosensor based on yfiA gene Khlebodarova T.M., Kachko A.V., Stepanenko I.L., Tikunova N.V. 5th International Conference on Bioinformatics of Genome Regulation and Structure (BGRS'2006), Novosibirsk, Russia (July 14-22) |
|
| Development of a computer system for the automated reconstruction of molecular-genetic interaction networks Aman E.E., Demenkov P.S., Pintus S.S., Nemiatov A.I., Apasieva N.V., Dubovenko E.A., Ignatieva E.V., Podkolodny N.L., Ivanisenko 5th International Conference on Bioinformatics of Genome Regulation and Structure (BGRS'2006), Novosibirsk, Russia (July 14-22) |
|
| FASTPROT: A computtational workbench for analysis of function, activity and structure of protein Ivanisenko V.A., Demenkov P.S., Fomin E.S., Oshurkov I.S., Korotkov R.O., Aman E.E., Pintus S.S., Startcev K.S., Tatarnikova L.Yu., Panina I.I., Sobolev A.A., Selenev I.V., Elohov V.J., Golovina A.N., Aknazarov Z.I., Sharonova I.V., Krestianova M.A., etc. 5th International Conference on Bioinformatics of Genome Regulation and Structure (BGRS'2006), Novosibirsk, Russia (July 14-22) |
|
| HCV-KINET database: kinetic parameter reactions and regulatory processes of the life cycle of the hepatitis C virus Mishchenko E.L., Korotkov R.О., Ivanisenko V.A. 5th International Conference on Bioinformatics of Genome Regulation and Structure (BGRS'2006), Novosibirsk, Russia (July 14-22) |
|
| PDBSite database and PDBSiteScan tool: recognition of functional sites in protein 3D structure and template-based docking Ivanisenko V.A., Ivanisenko T.V., Sharonova I.V., Krestyanova M.A., Ivanisenko N.V., Grigorovich D.A. 5th International Conference on Bioinformatics of Genome Regulation and Structure (BGRS'2006), Novosibirsk, Russia (July 14-22) |
|
| Prediction in changes of protein thermodynamic stability upon single mutations Demenkov P.S, Ivanisenko V.A. 5th International Conference on Bioinformatics of Genome Regulation and Structure (BGRS'2006), Novosibirsk, Russia (July 14-22) |
|
| Rise of new zn2+ binding sites can be a molecular mechanism for impaired function of the p53 mutants Fomin E.S., Oshurkov I.S., Ivanisenko V.A. 5th International Conference on Bioinformatics of Genome Regulation and Structure (BGRS'2006), Novosibirsk, Russia (July 14-22) |
|
| MGSmodeller - компьютерная система для конструирования, расчета и анализа моделей молекулярно-генетических систем Казанцев Ф.В. , Подколодная Н.Н., Акбердин И.Р., Безматерных К.Д., Крокус И.В., Лашин С.А., Ратушный А.В., Лихошвай В.А. 7 Всероссийская конференция молодых ученых по математическому моделированию и информационным технологиям, Красноярск, Россия, (Ноябрь 1-3) |
|
| System computer biology in Institute of Cytology of SB RAS Yu. Matushkin, E. Ananko, D. Afonnikov, V. Golomolzin, V. Ivanisenko, E. Ignat’eva, A. Katokhin, V.Likhoshvai, D. Miginskii, N. Omelyanchuk, S. Peltek, T. Khlebodarova, N. Kolchanov Saint-Petersburg International Workshop on NanoBiotechnologies |
|
| A minimum mathematical model for suppression HCV RNA replication in cell culture Безматерных К.Д., Мищенко Е.Л., Ратушный А.В., Лихошвай В.А., Хлебодарова Т.М., Иванисенко В.А. The 2d All-Russian Conference "Infocommunication and Computing Technologies and Systems", Ulan-Ude |
|
| Mathematical model for suppression of hepatitis C virus RNA replicon replication in cell culture Bezmaternikh K.D., Mishchenko E.L., Ratushny A.V., Likhoshvai V.A., Khlebodarova T.M., Ivanisenko V.A. The VII All-Russian young scientists conference on mathematical modeling and informatics technology (with foreign participants), Krasnoyarsk |
|
| Анализ распределения просайтов функциональных сайтов в пространственных структурах белков Шаронова (Медведева) И. В. XLIV Международная научная студенческая конференция "Студент и научно-технический прогресс" |
|
| Rise of new binding sites can be a molecular mechanism for impaired function of the p53 mutants Fomin E.S., Oshurkov I.S., Ivanisenko V.A 5th International Conference on Bioinformatics of Genome Regulation and Structure (BGRS'2006), Novosibirsk, Russia (July 14-22) |
|
| New Zn 2+ binding sites in the p53 mutants are promising targets for antitumor drug design Fomin E.S., Oshurkov I.S., Ivanisenko V.A 3th International Conference "Genomics, Proteomics, Bioinformatics and Nanotechnologies for Medicine" (GPBM 2006) |
|
| TRRD: a database on experimentally identified transcription regulatory regions and transcription factor binding sites. Kolchanov N.A., Podkolodnaya O.A., Ananko E.A., Ignatieva E.V., Stepanenko I.L., Khlebodarova T.M., Merkulov V.M., Merkulova T.I., Podkolodny N.L., Romashchenko A.G. 5th International Conference on Bioinformatics of Genome Regulation and Structure (BGRS'2006), Novosibirsk, Russia (July 14-22) |
|
| A minimum mathematical model for suppression HCV RNA replication in cell culture. Bezmaternikh K.D., Mishenko E.L., Likhoshvai V.A., Khlebodarova T.M., Ivanisenko V.A. 5th International Conference on Bioinformatics of Genome Regulation and Structure (BGRS'2006), Novosibirsk, Russia (July 14-22) |
|
| In silico cell II. Information sources for modeling Escherichia coli metabolic pathways. Khlebodarova T.M., Ananko E.A., Nedosekina (Oschepkova) E.A. Podkolodnaya O.A., Poplavsky A.S., Smirnova O.G., Ibragimova S.M., Mishenko E.L., Stepanenko I.L., Ignatieva E.V., Proskura A.L., Nishio Imaizumi, Usuda Matsui, Podkolodny N.L. 5th International Conference on Bioinformatics of Genome Regulation and Structure (BGRS'2006), Novosibirsk, Russia (July 14-22) |
|
| Сonstruction and structure analysis of apoptosis gene network in hepatitis C Stepanenko I.L., Podkolodnaya N.N., Paramonova N.V., Podkolodny N.L. Proceedings Of The Fifth International Conference On Bioinformatics Of Genome Regulation And Structure (BGRS'2006) |
|
| Mathematical modeling of the purine biosynthesis in Escherichia coli Nedosekina E., Likhoshvai V.A. Genomics, Proteomics, Bioinformatics and Nanotechnologies for Medicine |
|
| Mathematical modeling of regulation of Escherichia coli purine biosynthesis pathway enzymatic reactions Nedosekina E.A. Fifth international conference on bioinformatics of genome regulation and structure (BGRS'2006) |
|
| Modeling of the effects of threonine, valine, isoleucine and pyridoxal 5'-monophosphate on biosynthetic threonine dehydratase reaction Nedosekina E.A., Usuda Y., Matsui K. Fifth international conference on bioinformatics of genome regulation and structure (BGRS'2006) |
|
| Regulation of pyrimidine biosynthesis in Escherichia coli: gene network reconstruction and mathematical modeling Ratushny A.V., Nedosekina E.A. Fifth international conference on bioinformatics of genome regulation and structure (BGRS'2006) |
|
| Development of a computer system for the automated reconstruction of molecular-genetic interaction networks. Aman E.E., Demenkov P.S., Pintus S.S., Nemiatov A.I., Apasieva N.V., Dubovenko E.A., Ignatieva E.V., Podkolodny N.L., Ivanisenko V.A. Proceedings Of The Fifth International Conference On Bioinformatics Of Genome Regulation And Structure (BGRS'2006) |
|
| Компьютерные подходы к исследованию влияния полиморфизмов на экспрессию генов и функцию белков. Колчанов Н.А., Иванисенко В.А., Кочетов А.В., Игнатьева Е.В., Пономаренко М.П., Орлова Г.В., Меркулова Т.И., Ощепков Д.Ю., Аман Е.Э., Кузнецова Т.Н., Мордвинов В.А., Деменков П.С., Пинтус С.С., Ратушный А.В., Лихошвай В.А., Ананько Е.А., Подколодный Н.Л., Подколодная Н.Н., Турнаев И.И., Апасьева Н.В., Губина М.А., Ромащенко А.Г. International conference “Basic Science for Biotechnology and Medicine” September 3-7, 2006, Novosibirsk. Russia. |
|
| Bioinformatics and mechanisms of molecular pathology N.A. Kolchanov, V. Ivanisenko, M. Ponomarenko, E. Aman, E. Ignatieva, T. Kuznetsova, V. Mordvinov, P. Demenkov, A. Ratushny, V.Likhoshvai, E. Ananko, N. Podkolodny, N. Podkolodnaya, A.. Romaschenko Геномика, протеомика, биоинформатика и нанотехнологии для медицины – GPMB-2006 Новосибирск, июль 2006 |
|
| 2005 | Discrete Modeling of Structure Preserving Mutations in Proteins Pavel S. Demenkov, Evgeny Y. Kharlamov 9-th Asian Logic Conference |
| Modelling of signal transduction and gene expression regulation in immune system cells. Nedosekina E.A., Ananko E.A., Likhoshvai V.A., Oshchepkov D.Yu., Kolchanov N.A. Сonference on modeling and simulation in biology, medicine and biomedical engineering. Linkoping, Sweden, 2005, May 26-27 |
|
| 2004 | PDBSITE, PDBLIGAND and PDBSITESCAN: a computational workbench for the recognition of the structural and functional determinants in protein tertiary structures combined with protein draft docking Ivanisenko V.A., Pintus S.S., Krestyanova M.A., Demenkov P.S., Znobisheva E.K., Ivanov E.E., Grigorovich D.A. 2. Fourth Intern. Conf. on Bioinformatics of Genome Regulation and Structure |
| Разработка логико-вероятностной модели белков, определяющей структурную ограниченность белков Деменков П.С. 42-я МНСК «Студент и научно-технический прогресс» |
|
| 2003 | Emperical Theory Discovery. Деменков П.С., Витяев Е.Е. Вероятностные Идеи в Науке и Философии |
 Гранты
Гранты
| 2021 | Изучение межклеточной миграции ядер в мейозе высших растений:
трехмерный ультраструктурный анализ, иммуноокрашивание целых органов и
визуализация в живых клетках РФФИ, номер гранта 21-54-50001 |
| 2020 | Разработка персонализированных подходов к оценке вариабельности гликемии у больных сахарным диабетом 1 типа на основе математических методов и искусственного интеллекта Российский научный фонд, номер гранта 20-15-00057 |
| 2019 | Влияние мутаций на макроскопические свойства белков: теоретическое исследование механизмов с помощью методов молекулярного моделирования РФФИ, номер гранта 19-44-543002 |
| Эволюция эндосимбиотической бактерии Wolbachia и закономерность распространения симбионта среди насекомых РФФИ, номер гранта 19-04-00983 А |
|
| 2018 | Анализ дифференциальной экспрессии генов в отделах головного мозга крыс, селектированных по агрессивности РФФИ, номер гранта 18-34-00496 |
| 2017 | Оценочная функция ранжирования результатов молекулярного докинга и ее применение для дизайна ингибиторов дигидродипиколинат синтазы (DapA) из Mycobacterium tuberculosis и супероксиддисмутазы (SOD1), ассоциированной с боковым амиотрофическим склерозом человека РФФИ, номер гранта 17-54-49004 |
| 2016 | Популяционная структура и следы адаптации к холодному климату в геномах российских пород крупного рогатого скота и овец Российский научный фонд |
| 2013 | Разработка программного комплекса для анализа влияния SNP на функцию генов, связанных с развитием социально значимых заболеваний Минобрнауки, номер гранта ФЦП, ГК 07.514.13.0094 |
| 2012 | Изучение механизмов функционирования репликона вируса гепатита C в клетке хозяина методами системной биологии Министерство науки и образования России, номер гранта 11.519.11.6041 |
| Междисциплинарный интеграционный проект «Математические модели, числен-ные методы и параллельные алго-ритмы для решения больших задач СО РАН и их реализация на мно-гопроцессорных СуперЭВМ» Предизиум СО РАН, номер гранта № 130 |
|
| Междисциплинарный интеграционный проект "Исследование закономерностей и тенденций развития самоорганизующихся систем на примере веб-пространства и биологических сообществ" Президиум СО РАН, номер гранта № 21 |
|
| 2011 | Компьютерный анализ и моделирование процессов развития апикальной меристемы побега РФФИ, номер гранта 11-04-01748 |
| Проектирование и разработка RESTful-веб-сервисов для создания распределенной инфраструктуры, ориентированной на решения задач реконструкции и анализа генных сетей Министерство образования и науки, номер гранта 07.514.11.4023 |
|
| Масштабируемые параллельные программы молекулярной динамики для решения задач биотехнологии и компьютерной фармакологии Минобрнаука, номер гранта 07.514.11.4011 |
|
| Разработка алгоритмов и программных систем для решения задач анализа последовательностей, возникающих в теоретической и прикладной геномике Министерство образования и науки РФ, номер гранта 07.514.11.4003 |
|
| Выявление и анализ особенностей аминокислотных последовательностей нейраминидазы и гемагглютинина вируса гриппа человека, определяющих их адгезивные свойства РФФИ, номер гранта 11-04-92712 |
|
| 2010 | EU-FP7 Research Project SysPatho EU, номер гранта 260429 |
| Разработка алгоритмов для биоинформационного анализа комплексных метаболических и молекулярно-генетических сетей Министерство образования и науки, номер гранта 14.740.11.0001 |
|
| 2009 | Разработка Веб-ориентированной экспертной системы, предназначенной для распознавания взаимосвязанных белков, выявленных с применением постгеномных методов Федеральное агенство по науке и инновациям, номер гранта 02.514.11.4123 |
| Разработка программно-информационного комплекса для исследования механизмов регуляции экспрессии генов эукариот Министерство образования и науки, номер гранта П-721 |
|
| Биоинформатика генетической изменчивости: исследование влияния мутаций на молекулярно-генетические системы организмов Президиум РАН, номер гранта 23.29 |
|
| ПОСТГЕНОМНАЯ БИОИНФОРМАТИКА: КОМПЬЮТЕРНЫЙ АНАЛИЗ И МОДЕЛИРОВАНИЕ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКИХ СИСТЕМ Президиум СО РАН, номер гранта 119 |
|
| Поиск, селекция и изучение перспективных бактериальных штаммов-продуцентов для переработки глицерина Номер гранта программа РАН № 19.13 |
|
| 2008 | Создание баз данных в области нанобиотехнологии как элементов информационной составляющей инфраструктуры наноиндустрии ФАНИ, номер гранта № 01.647.11.2013 |
| Компьютерная реконструкция потенциальных регуляторных районов E.coli и механизмов их функционирования. Экспериментальное подтверждение разработанных моделей РФФИ, номер гранта 08-04-01008-а |
|
| 2007 | Разработка универсальных геносенсоров для детекции общей токсичности, мутагенности и техногенных стрессов ФАНИ, номер гранта №02.512.11.2165 |
| 2006 | Применение высоких давлений к исследованию молекулярных кристаллов СО РАН (интеграционный проект), номер гранта 49 |
| 2005 | Теоретическое исследование особенностей эволюции белков, обусловленных координированными заменами аминокислотных остатков РФФИ, номер гранта 05-04-49141 |
| Компьютерный анализ структурной организации и эволюции функциональных сайтов глобулярных белков РФФИ, номер гранта 05-04-49283 |
|
| 2004 | Protein Folding: A Kinetic Approach to the Mechanism CRDF, номер гранта CRDF Rup2-2629-NO-04 |
| 1999 | Экспериментальное и теоретическое исследование пространственной структуры и процесса ренатурации белковых молекул РФФИ, номер гранта 99-04-49879 |
 Научное руководство
Научное руководство
| 2013 | Компьютерный анализ связи конформационных пептидов с аллергенностью белков Брагин Анатолий Олегович , 2013-05-22 |
| 2011 | Предсказание изменения термодинамической стабильности белков при одиночных мутациях аминокислот Корепанова Ольга Александровна 2011-06-16 |
| Предсказание белок-белковых взаимодействий на основе аминокислотных последовательностей Оконечников Константин Вячеславович 2011-06-10 |
|
| 2010 | КОМПЬЮТЕРНЫЙ АНАЛИЗ СТРУКТУРЫ И ЭВОЛЮЦИИ ФУНКЦИОНАЛЬНЫХ САЙТОВ БЕЛКА P53 ПИНТУС СЕРГЕЙ СЕРГЕЕВИЧ , 2010-09-20 |
| Создание базы данных и компьютерный анализ химических соединений, потенциальных биомаркеров заболеваний Сайк Ольга Владимировна 2010-06-18 |
|
| 2004 | МАТЕМАТИЧЕСКАЯ МОДЕЛЬ ГЕННОЙ СЕТИ TNFalpha ИНДУЦИРУЕМОЙ АКТИВАЦИИ NF-kappaB Гурьева Я.П. , 2004-06-06 |
| 2000 | Генная сеть апоптоза Григорьев С.А. 2000-06-06 |
 Учебные курсы
Учебные курсы
| 2018 | Организация и функционирование молекулярно-генетических систем IV: генные сети (отв. за чтение курса - Игнатьева Е.В.) Игнатьева Е.В., Сайк О.В. НГУ, Кафедра информационной биологии ФЕН, семестров: 1 |
| 2017 | Организация и функционирование молекулярно-генетических систем IV: генные сети (отв. за чтение курса - Игнатьева Е.В.) Игнатьева Е.В., Сайк О.В. НГУ, Кафедра информационной биологии ФЕН, семестров: 1 |
| 2015 | Информационные технологии в биологии Деменков Павел Сергеевич Лекции и практические занятия для аспирантов ИЦиГ СО РАН по специальности «Математическая биология, биоинформатика», семестров: 1 |
| 2011 | Структурная компьютерная биология, лекции. НГУ, Факультет естественных наук, Кафедра информационной биологии, семестров: 1 |
| 2010 | Структурная компьютерная биология, лекции. НГУ, Факультет естественных наук, Кафедра информационной биологии, семестров: 1 |
| Методы анализа биологических данных НГУ, ММФ, Кафедра дискретной математики и информатики, семестров: 1 |
|
| 2009 | Структурная компьютерная биология, лекции. НГУ, Факультет естественных наук, Кафедра информационной биологии, семестров: 1 |
| 2008 | Структурная компьютерная биология, лекции. НГУ, Факультет естественных наук, Кафедра информационной биологии, семестров: 1 |
| 2007 | Структурная компьютерная биология, лекции. НГУ, Факультет естественных наук, Кафедра информационной биологии, семестров: 1 |
| 2006 | Структурная компьютерная биология, лекции. НГУ, Факультет естественных наук, Кафедра информационной биологии, семестров: 1 |
| 2004 | Генные сети НГУ, ФЕН, КИБ, семестров: 1 |
 Патенты
Патенты
| 2023 | Программа для предсказания взаимодействий между вершинами ассоциативной сети (ЭНДИнтерПредикт) / A program for predicting interactions between vertices of an associative network (ANDInterPredict) Иванисенко Тимофей Владимирович, Деменков Павел Сергеевич, Иванисенко Владимир Александрович Номер патента 2023680578 |
| Программа для генерации обучающих выборок, предназначенных для глубокого машинного обучения графовых нейронных сетей (ЭНДТрэйнингСет) / A program for generating training sets for deep machine learning of graph neural networks (ANDTrainingSet) Иванисенко Тимофей Владимирович, Деменков Павел Сергеевич, Иванисенко Владимир Александрович Номер патента 2023680472 |
|
| Программа для обучения графовой нейронной сети предсказанию ассоциативных связей между вершинами графа (ЭНДИнтерКоммон) / A program for deep machine learning of a graph neural network to predict associative interactions between vertices of a graph (ANDInterCommon) Иванисенко Тимофей Владимирович, Деменков Павел Сергеевич, Иванисенко Владимир Александрович Номер патента 2023681228 |
|
| 2022 | Способ определения индивидуального риска формирования плоскоклеточного рака легкого на основе анализа микробиома мокроты Дружинин В.Г., Минина В.И., Баранова Е.Д., Волобаев В.П., Мацкова Л.В., Деменков П.С., Ларионов А.В., Баканова М.Л. Номер патента 2771855 |
| Компьютерная программа генерации обучающих выборок для машинного обучения моделей классификации имён биологических объектов в неструктурированных текстах (ЭНДГен) / Computer program of the generating of learning sets for the training of models aimed on the classification of names of biological objects in unstructured texts (ANDGen) Иванисенко Тимофей Владимирович, Деменков Павел Сергеевич, Иванисенко Владимир Александрович Номер патента 2022668790 |
|
| Программа для обучения моделей классификации имён биологических объектов в размеченных текстах, на основе контекста (ЭНДТрэин) / A program for training of classification models for the classification of names of biological entities in the pre-mapped texts, on the basis of their context (ANDTrain) Иванисенко Тимофей Владимирович, Деменков Павел Сергеевич, Иванисенко Владимир Александрович Номер патента 2022667646 |
|
| Программа оценки точности разметки коротких имён биологических объектов в неструктурированных текстах на английском языке (ЭНДПред) / The program for assessing the accuracy of mapped short names of biological objects in the unstructured english texts (ANDPred) Иванисенко Тимофей Владимирович, Деменков Павел Сергеевич, Иванисенко Владимир Александрович Номер патента 2022668741 |
|
| 2021 | Программа для расчета значений совстречаемости пар биологических объектов из онтологии ЭНДСистем в текстах рефератов научных публикаций (ЭНДМатч) / Program for pairwise scoring of co-occurrences between biological objects from the ontology of ANDSystem in the unstructured texts of abstracts of scientific publications (ANDMatch) Иванисенко Владимир Александрович, Деменков Павел Сергеевич, Иванисенко Тимофей Владимирович Номер патента 2021664006 |
| Программа для парного структурного выравнивания третичных структур белков (ТСБ) / Program for pairwise structure alignment of tertiary structures of proteins by secondary structure elements and atomic coordinates (TSP) Иванисенко Владимир Александрович, Иванисенко Тимофей Владимирович, Иванисенко Никита Владимирович Номер патента 2021663894 |
|
| Биотехнологически значимые штаммы бактерий (МикроБиотех) / Biotechnologically significant strains of bacteria (MicroBiotech) Мухин А.М., Лахова Т.Н., Клименко А.И., Казанцев Ф.В., Деменков П.С., Лашин С.А. Номер патента 2021621155 |
|
| 2020 | База данных дайджестов научных публикаций в области биологии (ЭНДДайджестДБ) / Database of digests of scientific literature in the field of biology (ANDDigestDB) Иванисенко Тимофей Владимирович, Деменков Павел Сергеевич, Иванисенко Владимир Александрович Номер патента 2020621827 |
| Программный комплекс для управления веб ресурсом автоматической генерации дайджестов научных публикаций в области биологии (ЭНДДайджест) / Software package for the management of the web-based portal for the automated generation of digests of scientific publications in the field of biology (ANDDigest) Иванисенко ТВ, Деменков ПС, Иванисенко ВА Номер патента 2020660515 |
|
| Программа предсказания аминокислотных замен, модулирующих активность и стабильность, в белках семейства протеиназ К (ПАКЗАС/PAASAS) Иванисенко Н.В., Алемасов Н.А., Иванисенко В.А. Номер патента 2020617253 |
|
| 2019 | Программа для оценки обогащённости функциональными взаимодействиями экспериментальных наборов генов (ФанДжинНет)/Program to estimate enrichment of functional interactions in experimental gene sets (FunGeneNet) Тийс Евгений Сергеевич, Деменков Павел Сергеевич, Иванисенко Тимофей Владимирович, Иванисенко Владимир Александрович Номер патента 2018619847 |
| 2013 | Позиции аминокислот функциональных сайтов белков в экзонной структуре кодирующих генов (СайтЭкс)/Protein functional sites positions in exon structure of the coding genes (SitEx) Медведева Ирина Вадимовна, Деменков Павел Сергеевич, Иванисенко Владимир Александрович Номер патента 2013621254 |
| 2012 | Интегрированная база данных молекулярно-генетических систем (МГСБаза)/Integrated molecular genetic systems database (MGSBase) Подколодный Н.Л., Семенычев А.В., Рассказов Д.А., Деменков П.С., Подколодная Н.Н., Ананько Е.А., Игнатьева Е.В., Подколодная О.А. Номер патента 2012620886 |
| Программа для предсказания аллергенности белков с использованием конформационных пептидов (Аллпред)/ Program for the prediction of allergenicity of proteins using conformation peptides (Allpred) Брагин Анатолий Олегович, Деменков Павел Сергеевич,Иванисенко Владимир Александрович Номер патента 2012615590 |
|
| 2010 | Программа для классификации биологических текстов на английском языке (БиоТекстКласс) / The program for classification of biological texts in English (BioTextClass) Иванисенко В.А., Деменков П.С., Иванисенко Т.В. Номер патента 2010617828 |
| База данных нанобиоматериалов (НАНОБИОБАЗА)/Nanobiomaterial database (NANOBIOBASE) Иванисенко В.А., Деменков П.С., Иванисенко Т.В., Подколодный Н.Л., Хлебодарова Т.М., Игнатьева Е.В., Ананько Е.А., Подколодная О.А., Колчанов Н.А. Номер патента 2010620013 |
|
| База данных нанобиотехнологий (НАНОБИОТЕХБАЗЕ)/Nanobiotechnology database (NANOBIOTECHBASE) Иванисенко В.А., Деменков П.С., Иванисенко Т.В., Подколодный Н.Л., Хлебодарова Т.М., Игнатьева Е.В., Ананько Е.А., Подколодная О.А., Колчанов Н.А. Номер патента 2010620014 |
|
| 2009 | База данных: Матрицы функциональных сайтов связывания транскрипционных факторов про- и эукариот (RealSite) Хлебодарова Т.М., Степаненко И.Л., Игнатьева Е.В., Ананько Е.А., Подколодная О.А. Номер патента №2009620500 |
| 2008 | База данных ассоциативных сетей (ЭНДЦелл)/Associative network database (ANDCell) Иванисенко В.А., Деменков П.С., Яркова Е.Э. Номер патента 2008620439 |
| Программа для реконструкции, визуализации и анализа ассоциативных сетей (ЭНДВизио)/Program for reconstruction, visualization and analyze associative networks (ANDVisio) Иванисенко В.А., Деменков П.С., Яркова Е.Э. Номер патента 2008615929 |
|
| Компьютерная система для конструирования, расчета и анализа моделей молекулярно-генетических систем (МГСмоделлер)/ A computer system for reconsnruction/ Calculation and analysis matimatical models of molecular genetic system (MGSmodeller) Лихошвай В.А., Казанцев Ф.В., Акбердин И.Р., Безматерных К.Д. Лашин С.А., Подколодная Н.Н., Ратушный А.В Номер патента №2008612820 |
|
| 2007 | База данных: Экспрессия эукариотических генов (ЭуГенЭксп) / Eukaryotic Gene Expression Database (EuGenExp) Ананько Е.А., Игнатьева Е.В., Подколодная О.А., Степаненко И.Л., Хлебодарова Т.М., Подколодный Н.Л., Колчанов Н.А. Номер патента №2007620394 |
| База данных: Транскрипционный Фактор-Ген Млекопитающих взаимодействия (ТФГМВ)/ Transcription Factor- Mammalian Gene Interactions (TFMGI) Колчанов Н.А., Подколодная О.А., Подколодный Н.Л., Хлебодарова Т.М., Ананько Е.А., Степаненко И.Л., Игнатьева Е.В., Генаев М.А. Номер патента №2007620395 |
|
| База данных: Сайты связывания транскрипционных факторов (ТФСАЙТ)/ Transcription factor sites database (TFSITE). Степаненко И.Л., Ананько Е.А., Игнатьева Е.В., Подколодная О.А., Хлебодарова Т.М., Подколодный Н.Л., Колчанов Н.А. Номер патента №2007620396 |
|
| 2006 | База данных: Регуляторные районы генов прокариот (ProkaTEX) Хлебодарова Т.М., Ананько Е.А., Подколодная О.А., Поплавский А.С., Наумочкин А.Н., Смирнова О.Г., Степаненко И.Л., Ибрагимова С.М., Мищенко Е.Л., Игнатьева Е.В., Подколодный Н.Л., Колчанов Н.А. Номер патента №2006620257 |
| База кинетических данных (KiNET) Недосекина Е.А., Хлебодарова Т.М., Поплавский А.С., Смирнова О.Г., Подколодная О.А., Ананько Е.А., Ибрагимова С.М., Мищенко Е.Л., Игнатьева Е.В., Подколодный Н.Л., Колчанов Н.А. Номер патента №2006620209 |
|
| База данных: Гены-сенсоры (GenSensor) Хлебодарова Т.М., Степаненко И.Л., Подколодный Н.Л. Номер патента №2006620197 |
| © 2010-2026 ИЦиГ СО РАН. Все права защищены. |